我有一个制表符分隔的文件,其中包含区域和这些区域中发现的相应生物实体(我已经检查了67个,因此您可以说每个区域都被检查了这67个实体的存在或缺失以及它们的频率)。
我将所有这些数据以表格格式呈现。
以下是示例数据:
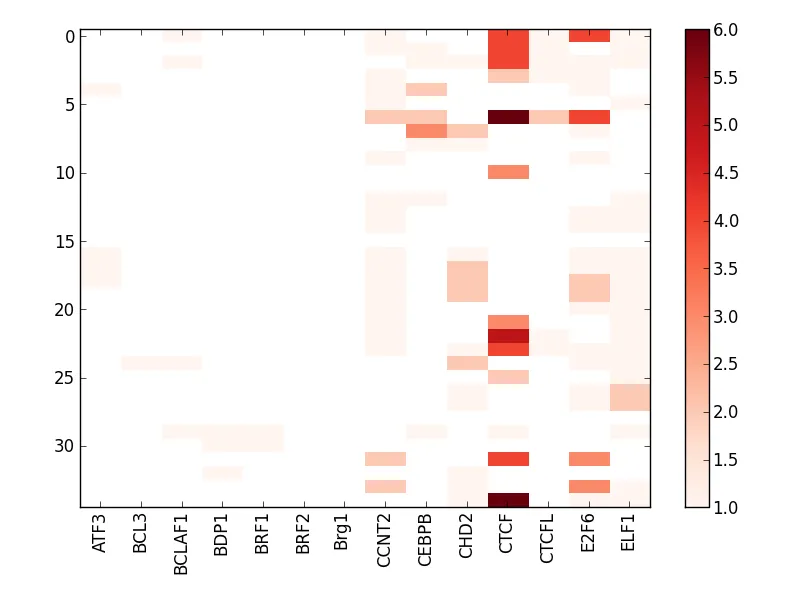
Region ATF3 BCL3 BCLAF1 BDP1 BRF1 BRF2 Brg1 CCNT2 CEBPB CHD2 CTCF CTCFL E2F6 ELF1
chr1:109102470:109102970 0 0 1 0 0 0 0 1 0 0 4 1 4 1
chr1:110526886:110527386 0 0 0 0 0 0 0 1 1 0 4 1 0 1
chr1:115300671:115301171 0 0 1 0 0 0 0 0 1 1 4 1 1 1
chr1:115323308:115323808 0 0 0 0 0 0 0 1 0 0 2 1 1 0
chr1:11795641:11796141 1 0 0 0 0 0 0 1 2 0 0 0 1 0
chr1:118148103:118148603 0 0 0 0 0 0 0 1 0 0 0 0 0 1
chr1:150521397:150521897 0 0 0 0 0 0 0 2 2 0 6 2 4 0
chr1:150601609:150602109 0 0 0 0 0 0 0 0 3 2 0 0 1 0
chr1:150602098:150602598 0 0 0 0 0 0 0 0 1 1 0 0 0 0
chr1:151119140:151119640 0 0 0 0 0 0 0 1 0 0 0 0 1 0
chr1:151128604:151129104 0 0 0 0 0 0 0 0 0 0 3 0 0 0
chr1:153517729:153518229 0 0 0 0 0 0 0 0 0 0 0 0 0 0
chr1:153962738:153963238 0 0 0 0 0 0 0 1 1 0 0 0 0 1
chr1:154155682:154156182 0 0 0 0 0 0 0 1 0 0 0 0 1 1
chr1:154155725:154156225 0 0 0 0 0 0 0 1 0 0 0 0 1 1
chr1:154192154:154192654 0 0 0 0 0 0 0 0 0 0 0 0 0 0
chr1:154192824:154193324 1 0 0 0 0 0 0 1 0 1 0 0 1 1
chr1:154192943:154193443 1 0 0 0 0 0 0 1 0 2 0 0 1 1
chr1:154193273:154193773 1 0 0 0 0 0 0 1 0 2 0 0 2 1
chr1:154193313:154193813 0 0 0 0 0 0 0 1 0 2 0 0 2 1
chr1:155904188:155904688 0 0 0 0 0 0 0 1 0 0 0 0 1 1
chr1:155947966:155948466 0 0 0 0 0 0 0 1 0 0 3 0 0 1
chr1:155948336:155948836 0 0 0 0 0 0 0 1 0 0 5 1 0 1
chr1:156023516:156024016 0 0 0 0 0 0 0 1 0 1 4 1 1 1
chr1:156024016:156024516 0 1 1 0 0 0 0 0 0 2 0 0 1 1
chr1:156163229:156163729 0 0 0 0 0 0 0 0 0 0 2 0 0 1
chr1:160990902:160991402 0 0 0 0 0 0 0 0 0 1 0 0 1 2
chr1:160991133:160991633 0 0 0 0 0 0 0 0 0 1 0 0 1 2
chr1:161474704:161475204 0 0 0 0 0 0 0 0 0 0 0 0 0 0
chr1:161509530:161510030 0 0 1 1 1 0 0 0 1 0 1 0 0 1
chr1:161590964:161591464 0 0 0 1 1 0 0 0 0 0 0 0 0 0
chr1:169075446:169075946 0 0 0 0 0 0 0 2 0 0 4 0 3 0
chr1:17053279:17053779 0 0 0 1 0 0 0 0 0 1 0 0 0 0
chr1:1709909:1710409 0 0 0 0 0 0 0 2 0 1 0 0 3 1
chr1:1710297:1710797 0 0 0 0 0 0 0 0 0 1 6 0 1 1
现在我该如何将这个数据用热力图表示,颜色从浅红到深红(根据频率),没有数据的地方用白色表示?
还有其他更好的方式来呈现这种类型的数据吗?
 如果您想要y轴的其他刻度,请使用
如果您想要y轴的其他刻度,请使用
plt.savefig("output.png")或类似方法),这样你的1100行才会有意义。 - Thorsten Kranz