levelplot()或基本图形中的image()的视觉灵活性与基本的heatmap()、pheatmap的pheatmap()或gplots的heatmap.2()的轻松聚类相结合。我想改变的是一个小细节——x轴标签的对角线方向。让我在代码中展示给你看。#example data
d <- matrix(rnorm(25), 5, 5)
colnames(d) = paste("bip", 1:5, sep = "")
rownames(d) = paste("blob", 1:5, sep = "")
levelplot()轻松地将方向更改为对角线:require(lattice)
levelplot(d, scale=list(x=list(rot=45)))
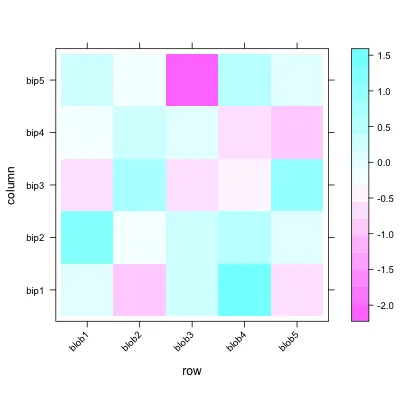
但是应用聚类似乎很麻烦。其他视觉选项,如在热图单元格周围添加边框,也一样。
现在,转向实际的heatmap()相关函数,聚类和所有基本的可视化都非常简单- 几乎不需要调整:
heatmap(d)
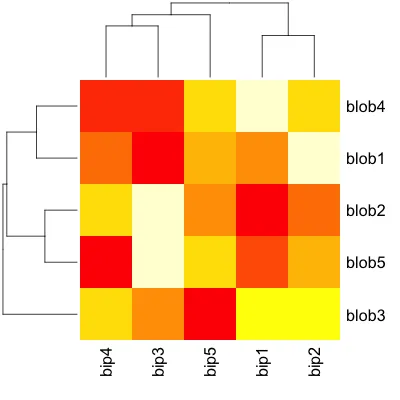
这里也是一样的:
require(gplots)
heatmap.2(d, key=F)
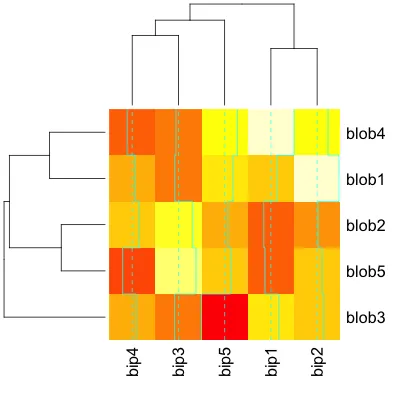
最后,我的最爱:
require(pheatmap)
pheatmap(d)
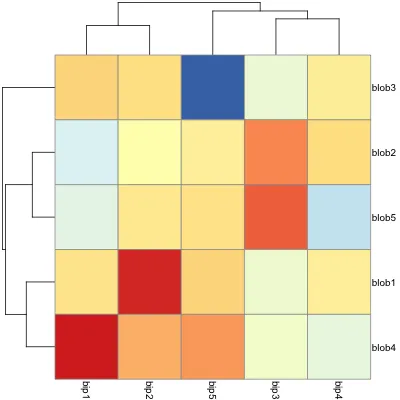
但是所有这些都没有旋转标签的选项。 pheatmap 的手册建议我使用grid.text来自定义我的标签方向。这真是太棒了,尤其是在聚类和更改显示标签顺序时。除非我在这里漏掉了什么......
最后,还有一个古老而好用的 image()。我可以旋转标签,它是最可定制的解决方案,但没有聚类选项。
image(1:nrow(d),1:ncol(d), d, axes=F, ylab="", xlab="")
text(1:ncol(d), 0, srt = 45, labels = rownames(d), xpd = TRUE)
axis(1, label=F)
axis(2, 1:nrow(d), colnames(d), las=1)
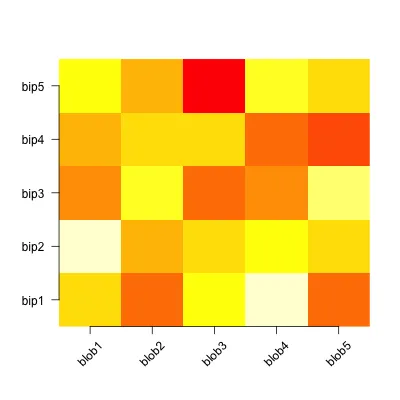
那么,我该怎么做才能获得我的理想热力图,包括聚类、方向和美观的可视化特征?我最好的办法是以某种方式改变 heatmap() 或 pheatmap(),因为这两者似乎在调整方面最为灵活。但欢迎任何解决方案。
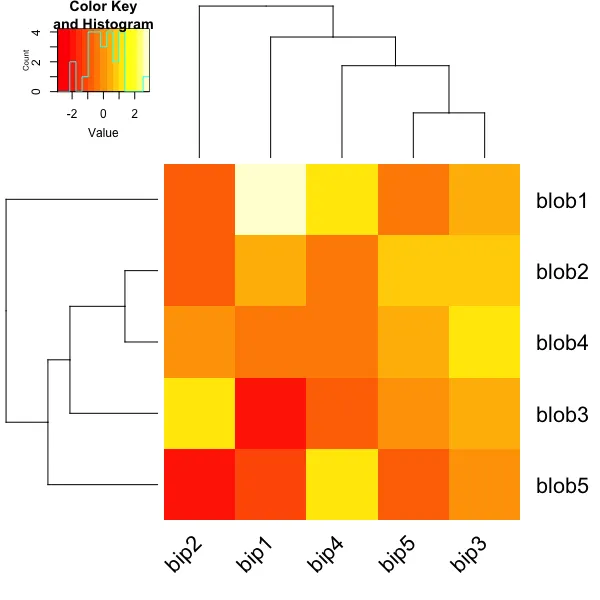
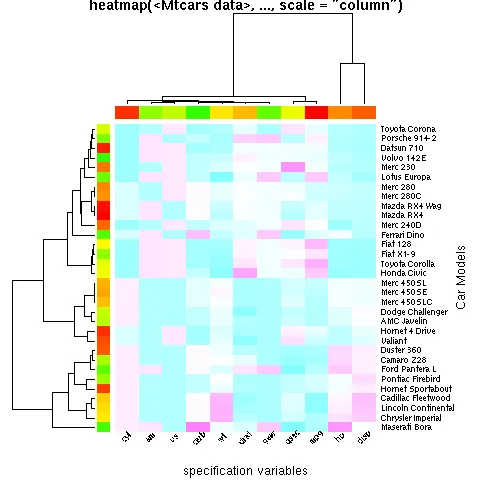
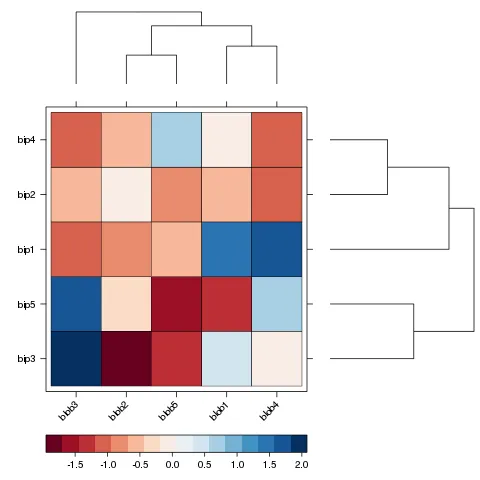
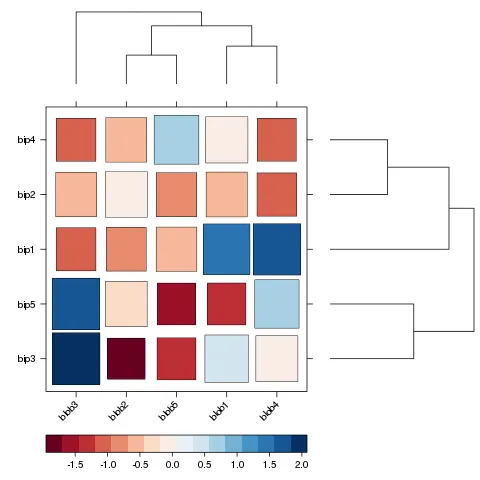
heatmap()是使用基本图形构建的,而不是像pheatmap()一样使用网格。 - Geek On Acid