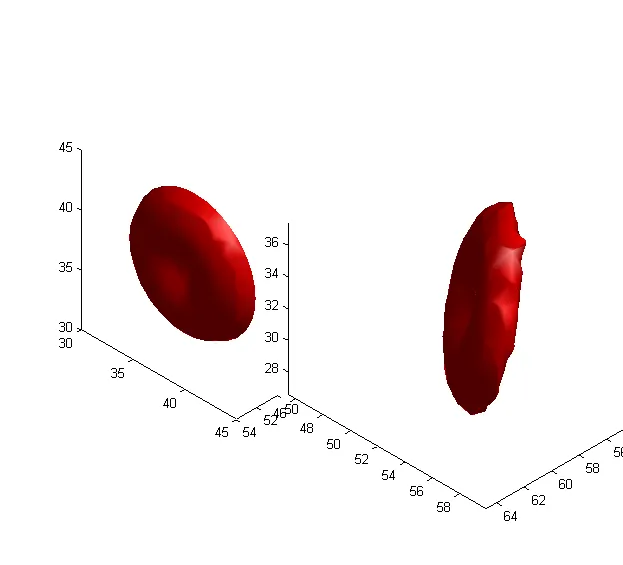
我有一个与超声数据对应的3D灰度体积。在Matlab中,这个3D体积仅是一个MxNxP的3D矩阵。我感兴趣的结构不是沿着z轴定向的,而是沿着一个已知的本地坐标系(x'y'z')定向的。到目前为止,我所拥有的类似于下图所示的东西,描述了原始的(xyz)坐标系和本地坐标系(x'y'z'):
[image]
我想通过本地坐标系上的特定平面获得该体积的2D投影(即图像),比如在z'=z0处。我该如何做?
如果该体积沿z轴定向,这个投影可以很容易地实现。也就是说,如果该体积在Matlab中被表示为V,则:
因此,投影可以通过计算数组沿第三维的总和来计算。但是,如果方向改变,问题就变得更加复杂。
我一直在研究Radon变换(2D,仅适用于2D图像而不是体积),并且也考虑了正交投影,但是目前我对该怎么做毫无头绪!
感谢任何帮助!
[image]
我想通过本地坐标系上的特定平面获得该体积的2D投影(即图像),比如在z'=z0处。我该如何做?
如果该体积沿z轴定向,这个投影可以很容易地实现。也就是说,如果该体积在Matlab中被表示为V,则:
projection = sum(V,3);
因此,投影可以通过计算数组沿第三维的总和来计算。但是,如果方向改变,问题就变得更加复杂。
我一直在研究Radon变换(2D,仅适用于2D图像而不是体积),并且也考虑了正交投影,但是目前我对该怎么做毫无头绪!
感谢任何帮助!