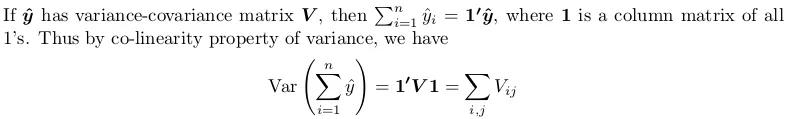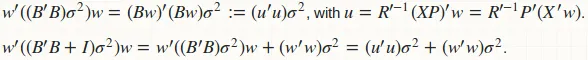您需要获取完整的方差-协方差矩阵,然后将其所有元素相加。 这是一个小证明:
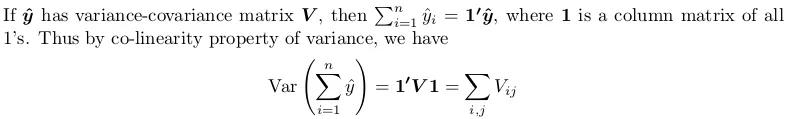
这里的证明使用了另一个定理,你可以在
Covariance-wikipedia中找到它:

具体来说,我们采用的线性变换是一个全为1的列矩阵。得到的二次型
计算方法如下,其中所有的
x_i和
x_j都是1。

设置
lm.tree <- lm(Volume ~ poly(Girth, 2), data = trees)
newdat <- data.frame(Girth = c(10, 12, 14, 16))
重新实现predict.lm以计算方差协方差矩阵
请参见如何使用predict.lm()计算置信区间和预测区间?了解predict.lm的工作原理。以下小函数lm_predict模仿它的工作方式,除了:
- 它不构建置信区间或预测区间(但是根据该Q&A中所述非常简单);
- 如果
diag = FALSE,它可以计算预测值的完整方差协方差矩阵;
- 它返回方差(对于预测值和残差),而不是标准误差;
- 它无法执行
type =“terms”; 它只能预测响应变量。
lm_predict <- function (lmObject, newdata, diag = TRUE) {
if (!inherits(lmObject, "lm")) stop("'lmObject' is not a valid 'lm' object!")
tm <- delete.response(terms(lmObject))
Xp <- model.matrix(tm, newdata)
pred <- c(Xp %*% coef(lmObject))
QR <- lmObject$qr
piv <- QR$pivot
r <- QR$rank
if (is.unsorted(piv)) {
B <- forwardsolve(t(QR$qr), t(Xp[, piv]), r)
} else {
B <- forwardsolve(t(QR$qr), t(Xp), r)
}
sig2 <- c(crossprod(residuals(lmObject))) / df.residual(lmObject)
if (diag) {
VCOV <- colSums(B ^ 2) * sig2
} else {
VCOV <- crossprod(B) * sig2
}
list(fit = pred, var.fit = VCOV, df = lmObject$df.residual, residual.var = sig2)
}
我们可以将其输出与
predict.lm 进行比较:
predict.lm(lm.tree, newdat, se.fit = TRUE)
lm_predict(lm.tree, newdat)
特别是:
oo <- lm_predict(lm.tree, newdat, FALSE)
oo
请注意,方差-协方差矩阵并不是以天真的方式计算的:
Xp %*% vcov(lmObject) % t(Xp),这种方法很慢。
聚合(求和)
在您的情况下,聚合操作是
oo$fit中所有值的总和。此聚合的均值和方差为:
sum_mean <- sum(oo$fit) ## mean of the sum
# 111.512
sum_variance <- sum(oo$var.fit) ## variance of the sum
# 6.671575
你可以使用t分布和模型中的剩余自由度,对这个汇总值进一步构建置信区间(CI)。
alpha <- 0.95
Qt <- c(-1, 1) * qt((1 - alpha) / 2, lm.tree$df.residual, lower.tail = FALSE)
sum_mean + Qt * sqrt(sum_variance)
构建预测区间(PI)需要进一步考虑残差方差。
VCOV_adj <- with(oo, var.fit + diag(residual.var, nrow(var.fit)))
sum_variance_adj <- sum(VCOV_adj)
sum_mean + Qt * sqrt(sum_variance_adj)
聚合(一般情况下)
一般的聚合操作可以是 oo$fit 的线性组合:
w[1] * fit[1] + w[2] * fit[2] + w[3] * fit[3] + ...
例如,求和操作的所有权重都为1;平均操作的所有权重都为0.25(在4个数据的情况下)。这是一个函数,它接受一个权重向量、一个显著性水平和由
lm_predict 返回的内容,以生成聚合统计信息。
agg_pred <- function (w, predObject, alpha = 0.95) {
if (length(w) != length(predObject$fit)) stop("'w' has wrong length!")
if (!is.matrix(predObject$var.fit)) stop("'predObject' has no variance-covariance matrix!")
agg_mean <- c(crossprod(predObject$fit, w))
agg_variance <- c(crossprod(w, predObject$var.fit %*% w))
VCOV_adj <- with(predObject, var.fit + diag(residual.var, nrow(var.fit)))
agg_variance_adj <- c(crossprod(w, VCOV_adj %*% w))
Qt <- c(-1, 1) * qt((1 - alpha) / 2, predObject$df, lower.tail = FALSE)
NAME <- c("lower", "upper")
CI <- setNames(agg_mean + Qt * sqrt(agg_variance), NAME)
PI <- setNames(agg_mean + Qt * sqrt(agg_variance_adj), NAME)
list(mean = agg_mean, var = agg_variance, CI = CI, PI = PI)
}
之前的求和操作进行了快速测试:
agg_pred(rep(1, length(oo$fit)), oo)
并进行平均操作的快速测试:
agg_pred(rep(1, length(oo$fit)) / length(oo$fit), oo)
备注
本答案已经改进,提供易于使用的功能,用于使用`lm()`进行线性回归:预测聚合预测值的预测区间。
升级(针对大数据)
太好了!非常感谢!有一件事我忘了提:在我的实际应用中,我需要对约300,000个预测值求和,这将创建一个大小约为700GB的完整方差-协方差矩阵。您是否知道是否有更加计算高效的方法直接得到方差-协方差矩阵的总和?
感谢使用`lm()`进行线性回归:聚合预测值的预测区间的原始帖子作者提供此非常有用的评论。是的,这是可能的,而且也(显著地)计算成本更低。目前,lm_predict形成方差-协方差矩阵如下:

agg_pred 函数计算预测方差(用于构建置信区间)为二次型: w'(B'B)w,并且计算预测方差(用于构建预测区间)为另一个二次型 w'(B'B + D)w,其中 D 是残差方差的对角矩阵。显然,如果我们合并这两个函数,就可以得到更好的计算策略:
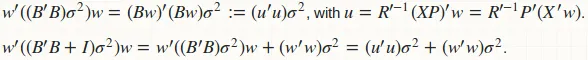
避免了对
B和
B'B的计算;我们将所有矩阵-矩阵乘法替换为矩阵-向量乘法。没有
B和
B'B的内存存储;只有
u,它只是一个向量。这是融合实现。
fast_agg_pred <- function (w, lmObject, newdata, alpha = 0.95) {
if (!inherits(lmObject, "lm")) stop("'lmObject' is not a valid 'lm' object!")
if (!is.data.frame(newdata)) newdata <- as.data.frame(newdata)
if (length(w) != nrow(newdata)) stop("length(w) does not match nrow(newdata)")
tm <- delete.response(terms(lmObject))
Xp <- model.matrix(tm, newdata)
pred <- c(Xp %*% coef(lmObject))
agg_mean <- c(crossprod(pred, w))
sig2 <- c(crossprod(residuals(lmObject))) / df.residual(lmObject)
QR <- lmObject$qr
piv <- QR$pivot
r <- QR$rank
u <- forwardsolve(t(QR$qr), c(crossprod(Xp, w))[piv], r)
agg_variance <- c(crossprod(u)) * sig2
agg_variance_adj <- agg_variance + c(crossprod(w)) * sig2
Qt <- c(-1, 1) * qt((1 - alpha) / 2, lmObject$df.residual, lower.tail = FALSE)
NAME <- c("lower", "upper")
CI <- setNames(agg_mean + Qt * sqrt(agg_variance), NAME)
PI <- setNames(agg_mean + Qt * sqrt(agg_variance_adj), NAME)
list(mean = agg_mean, var = agg_variance, CI = CI, PI = PI)
}
让我们进行快速测试。
fast_agg_pred(rep(1, nrow(newdat)), lm.tree, newdat)
fast_agg_pred(rep(1, nrow(newdat)) / nrow(newdat), lm.tree, newdat)
是的,答案正确!