多组密度图的交集
3
- Panda
2
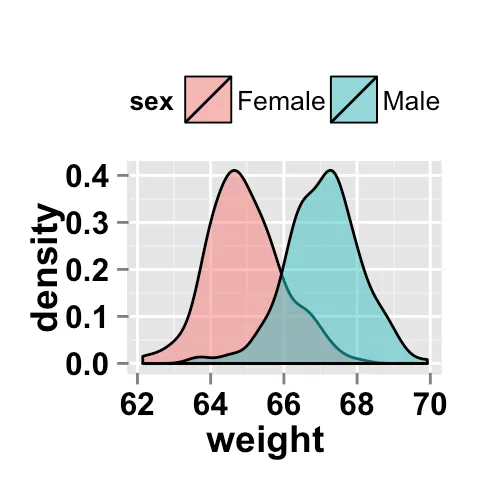
如果您对某个度量的群体差异感兴趣(在链接的图像中,它将是“重量”),那么为什么不直接进行t检验呢? - Chris Watson
根据您的数据需求,您似乎更需要咨询一位统计学家而不是程序员。如果您的问题是关于寻找统计上适当的测试或估计方法,那么您应该在[stats.se]上提问。如果您知道要执行哪个测试,但不知道如何在R中执行它,则应编辑您的问题以使其更清晰明了。 - MrFlick
2个回答
6
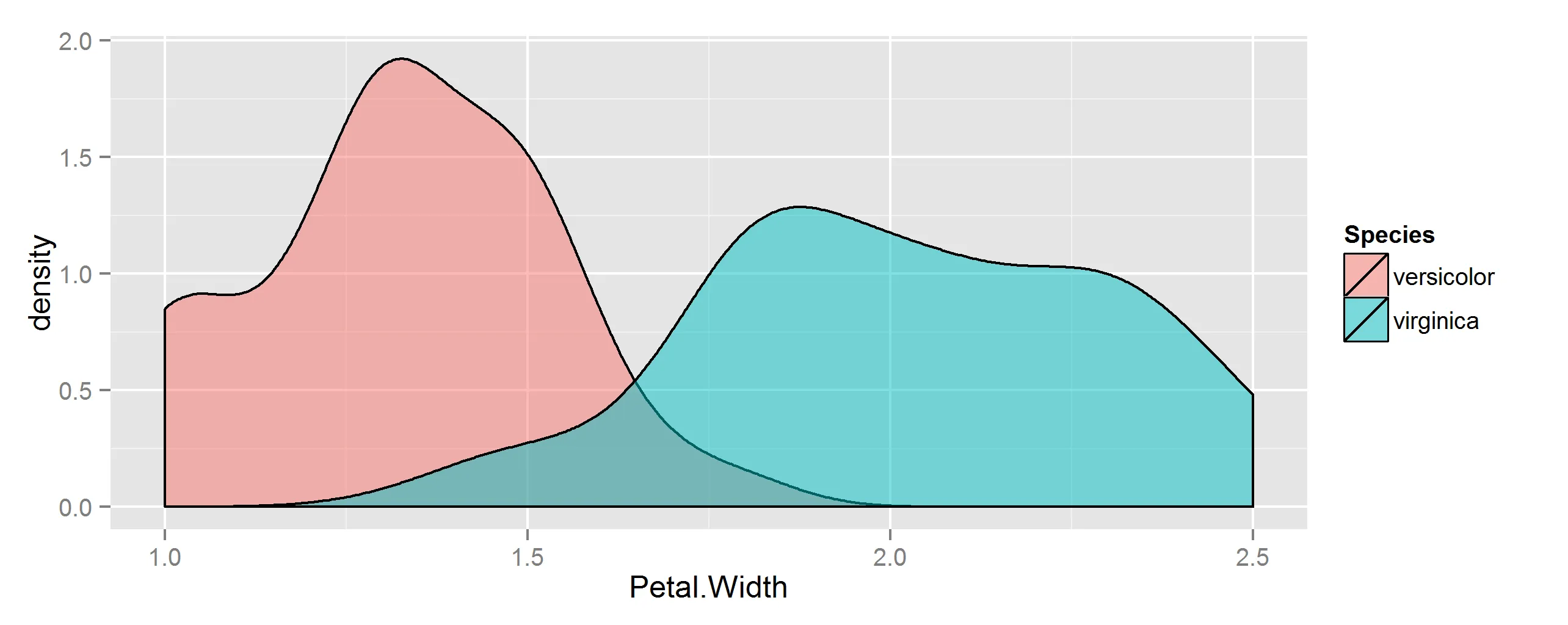
首先,准备一些数据以供使用。这里,我们将查看内置的iris数据集中两个植物物种的花瓣宽度。
## Some sample data from iris
dat <- droplevels(with(iris, iris[Species %in% c("versicolor", "virginica"), ]))
## make a similar graph
library(ggplot2)
ggplot(dat, aes(Petal.Width, fill=Species)) +
geom_density(alpha=0.5)
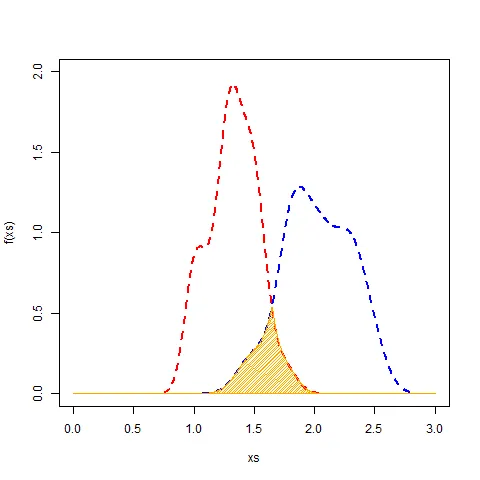
要找到交集的区域,您可以使用approxfun逼近描述重叠部分的函数。 然后对其进行积分以得到该区域的面积。 由于它们是密度曲线,因此它们的面积为1(左右),因此积分将是重叠百分比。
## Get density curves for each species
ps <- lapply(split(dat, dat$Species), function(x) {
dens <- density(x$Petal.Width)
data.frame(x=dens$x, y=dens$y)
})
## Approximate the functions and find intersection
fs <- sapply(ps, function(x) approxfun(x$x, x$y, yleft=0, yright=0))
f <- function(x) fs[[1]](x) - fs[[2]](x) # function to minimize (difference b/w curves)
meet <- uniroot(f, interval=c(1, 2))$root # intersection of the two curves
## Find overlapping x, y values
ps1 <- is.na(cut(ps[[1]]$x, c(-Inf, meet)))
ps2 <- is.na(cut(ps[[2]]$x, c(Inf, meet)))
shared <- rbind(ps[[1]][ps1,], ps[[2]][ps2,])
## Approximate function of intersection
f <- with(shared, approxfun(x, y, yleft=0, yright=0))
## have a look
xs <- seq(0, 3, len=1000)
plot(xs, f(xs), type="l", col="blue", ylim=c(0, 2))
points(ps[[1]], col="red", type="l", lty=2, lwd=2)
points(ps[[2]], col="blue", type="l", lty=2, lwd=2)
polygon(c(xs, rev(xs)), y=c(f(xs), rep(0, length(xs))), col="orange", density=40)
## Integrate it to get the value
integrate(f, lower=0, upper=3)$value
# [1] 0.1548127
- Rorschach
4
我喜欢之前的回答,但这个解释可能更易懂,同时我确保使用了一个通用的带宽:
library ( "caTools" )
# Extract common bandwidth
Bw <- ( density ( iris$Petal.Width ))$bw
# Get iris data
Sample <- with ( iris, split ( Petal.Width, Species ))[ 2:3 ]
# Estimate kernel densities using common bandwidth
Densities <- lapply ( Sample, density,
bw = bw,
n = 512,
from = -1,
to = 3 )
# Plot
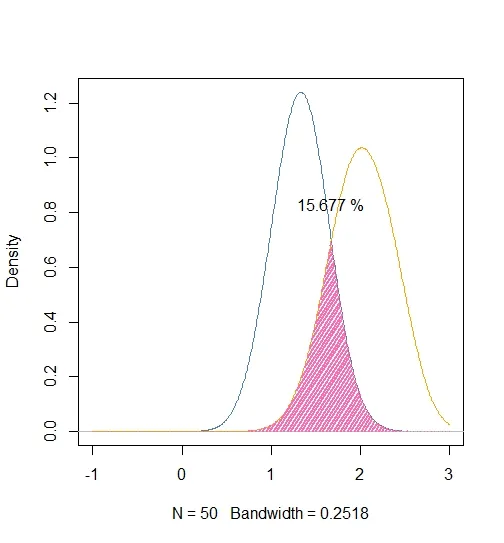
plot( Densities [[ 1 ]], xlim = c ( -1, 3 ),
col = "steelblue",
main = "" )
lines ( Densities [[ 2 ]], col = "orange" )
# Overlap
X <- Densities [[ 1 ]]$x
Y1 <- Densities [[ 1 ]]$y
Y2 <- Densities [[ 2 ]]$y
Overlap <- pmin ( Y1, Y2 )
polygon ( c ( X, X [ 1 ]), c ( Overlap, Overlap [ 1 ]),
lwd = 2, col = "hotpink", border = "n", density = 20)
# Integrate
Total <- trapz ( X, Y1 ) + trapz ( X, Y2 )
(Surface <- trapz ( X, Overlap ) / Total)
SText <- paste ( sprintf ( "%.3f", 100*Surface ), "%" )
text ( X [ which.max ( Overlap )], 1.2 * max ( Overlap ), SText )
- David.
2
好的答案+1,
pmin显然要简单得多!而且trapz是一个很酷的函数。不确定为什么带宽需要相同? - Rorschach谢谢!只有一个备注,我不应该将交集面积乘以2才能得到正确的比率吗?
例如,如果我有两个完全相等的PDF文件,它应该给出100%。但是,将交集区域的面积除以每个PDF区域的总和仅会让我得到50%。我错过了什么吗? - Panda
网页内容由stack overflow 提供, 点击上面的可以查看英文原文,
原文链接
原文链接