我想绘制一个2D核密度估计图。在这里,我发现seaborn包非常有用。然而,在长时间的搜索后,我仍然搞不清楚如何使y轴和x轴不透明,并显示等高线上的密度值。如果有人能够帮助我解决问题,我将不胜感激。请看下面我的代码和图表。 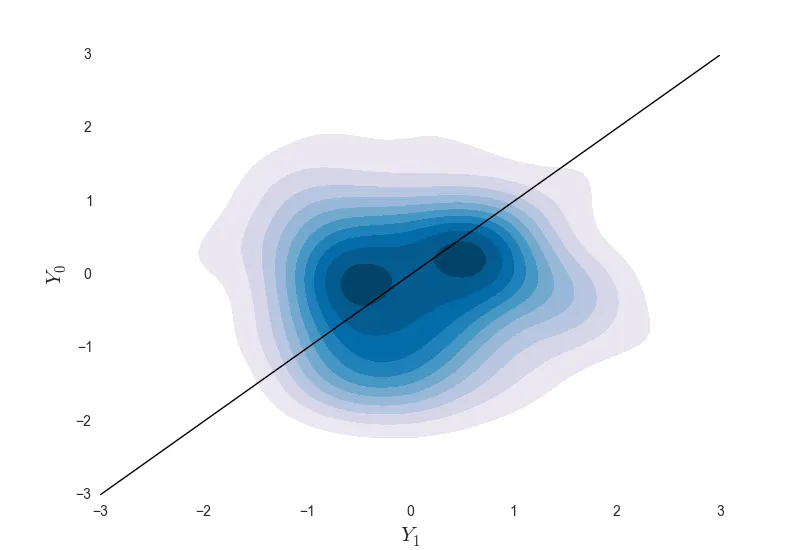
import numpy as np
import seaborn as sns
import matplotlib.pyplot as pl
Y = np.random.multivariate_normal((0, 0), [[0.8, 0.05], [0.05, 0.7]], 100)
ax = sns.kdeplot(Y, shade = True, cmap = "PuBu")
ax.patch.set_facecolor('white')
ax.collections[0].set_alpha(0)
ax.set_xlabel('$Y_1$', fontsize = 15)
ax.set_ylabel('$Y_0$', fontsize = 15)
pl.xlim(-3, 3)
pl.ylim(-3, 3)
pl.plot([-3, 3], [-3, 3], color = "black", linewidth = 1)
pl.show()
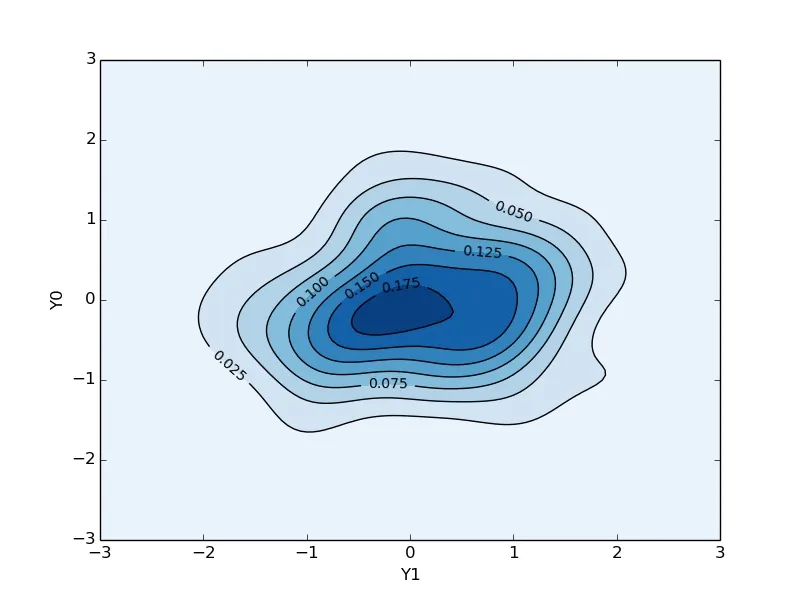
ax.collections[0].set_alpha(0)这行代码会使最低的等高线变成透明的;如果你不想这样,就不要包含这行代码。 - mwaskom