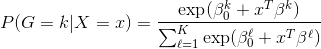您怎样从一个多项式模型的
下面是一个例子:
当我运行这个时,我得到了:
cv.glmnet对象中提取与特定的lambda相对应的系数呢?当我尝试使用二项式模型的语法进行操作时,coef函数返回的是系数稀疏矩阵的列表,而不是某个特定的稀疏矩阵。下面是一个例子:
tempcv <- cv.glmnet(x=as.matrix(iris[,-5]), y=iris[,5], family="multinomial",
nfolds=20, alpha=0.5)
coefsMin <- coef(tempcv, s="lambda.min")
当我运行这个时,我得到了:
> coefsMin[[3]]
5 x 1 sparse Matrix of class "dgCMatrix"
1
(Intercept) -19.091925
Sepal.Length .
Sepal.Width -3.755938
Petal.Length 4.355219
Petal.Width 8.909600
> coefsMin[[2]]
5 x 1 sparse Matrix of class "dgCMatrix"
1
(Intercept) 4.616488
Sepal.Length 1.649614
Sepal.Width .
Petal.Length -1.088160
Petal.Width -1.884997
> coefsMin[[1]]
5 x 1 sparse Matrix of class "dgCMatrix"
1
(Intercept) 14.475437
Sepal.Length -1.843070
Sepal.Width 5.312490
Petal.Length -2.698684
Petal.Width -5.708280
因此,coefsMin列表的条目可以有不同的系数水平和稀疏度。所有的系数集是否都与相同的lambda值对应?如果是,除了稀疏性之外,是否有选择其中一个的原因?
谢谢!