你可以使用scipy.interpolate.griddata和掩码数组,你可以使用参数method选择喜欢的插值类型,通常为'cubic',会做得非常好:
import numpy as np
from scipy import interpolate
array = np.random.random_integers(0,10,(10,10)).astype(float)
array[array>7] = np.nan
使用plt.imshow(array, interpolation='nearest')可以得到如下类似的结果:
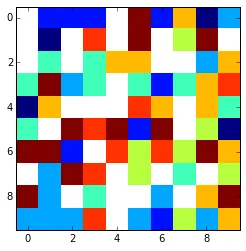
x = np.arange(0, array.shape[1])
y = np.arange(0, array.shape[0])
array = np.ma.masked_invalid(array)
xx, yy = np.meshgrid(x, y)
x1 = xx[~array.mask]
y1 = yy[~array.mask]
newarr = array[~array.mask]
GD1 = interpolate.griddata((x1, y1), newarr.ravel(),
(xx, yy),
method='cubic')
这是最终结果:
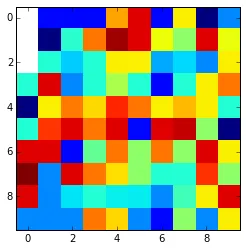
如果NaN值在边缘并被NaN值包围,则它们无法进行插值并将保持为nan。您可以使用fill_value参数更改它。
如果有一个3x3的NaN值区域,中间点能得到合理的数据吗?
这取决于您的数据类型,您需要进行一些测试。例如,您可以故意屏蔽一些良好的数据,尝试不同种类的插值,如立方体、线性等,并使用具有屏蔽值的数组计算插值值与先前屏蔽的原始值之间的差异,看看哪种方法返回最小差异。
您可以使用类似以下的代码:
reference = array[3:6,3:6].copy()
array[3:6,3:6] = np.nan
method = ['linear', 'nearest', 'cubic']
for i in method:
GD1 = interpolate.griddata((x1, y1), newarr.ravel(),
(xx, yy),
method=i)
meandifference = np.mean(np.abs(reference - GD1[3:6,3:6]))
print ' %s interpolation difference: %s' %(i,meandifference )
那就变成了这样:
linear interpolation difference: 4.88888888889
nearest interpolation difference: 4.11111111111
cubic interpolation difference: 5.99400137377
当然,这是随机数,结果可能会有很大差异。因此,最好的方法是在数据集的“有目的掩码”部分进行测试,看看会发生什么。


griddata接近我所寻找的内容。 - M.T