现在有一个基于HDF5的pickle克隆版本,名为hickle!
https://github.com/telegraphic/hickle
import hickle as hkl
data = {'name': 'test', 'data_arr': [1, 2, 3, 4]}
hkl.dump(data, 'new_data_file.hkl')
data2 = hkl.load('new_data_file.hkl')
print(data == data2)
编辑:
还有一种方法可以直接将数据“拨下来”,并压缩成一个档案,具体做法如下:
import pickle, gzip, lzma, bz2
pickle.dump(data, gzip.open('data.pkl.gz', 'wb'))
pickle.dump(data, lzma.open('data.pkl.lzma', 'wb'))
pickle.dump(data, bz2.open('data.pkl.bz2', 'wb'))
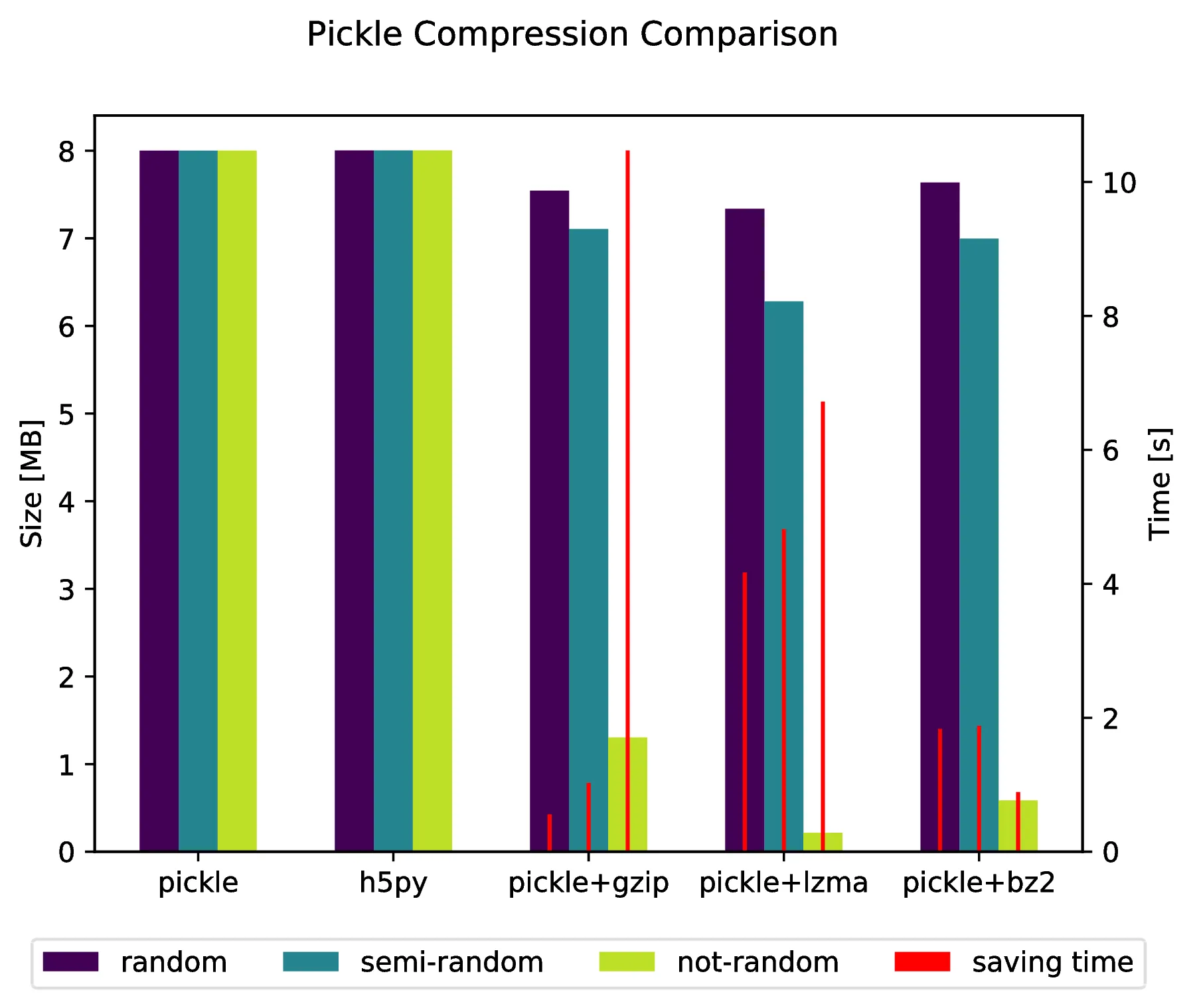
附录
import numpy as np
import matplotlib.pyplot as plt
import pickle, os, time
import gzip, lzma, bz2, h5py
compressions = ['pickle', 'h5py', 'gzip', 'lzma', 'bz2']
modules = dict(
pickle=pickle, h5py=h5py, gzip=gzip, lzma=lzma, bz2=bz2
)
labels = ['pickle', 'h5py', 'pickle+gzip', 'pickle+lzma', 'pickle+bz2']
size = 1000
data = {}
data['random'] = np.random.random((size, size))
data['semi-random'] = np.zeros((size, size))
for i in range(size):
for j in range(size):
data['semi-random'][i, j] = np.sum(
data['random'][i, :]) + np.sum(data['random'][:, j]
)
data['not-random'] = np.arange(
size * size, dtype=np.float64
).reshape((size, size))
sizes = {}
for key in data:
sizes[key] = {}
for compression in compressions:
path = 'data.pkl.{}'.format(compression)
if compression == 'pickle':
time_start = time.time()
pickle.dump(data[key], open(path, 'wb'))
time_tot = time.time() - time_start
sizes[key]['pickle'] = (
os.path.getsize(path) * 10**-6,
time_tot.
)
os.remove(path)
elif compression == 'h5py':
time_start = time.time()
with h5py.File(path, 'w') as h5f:
h5f.create_dataset('data', data=data[key])
time_tot = time.time() - time_start
sizes[key][compression] = (os.path.getsize(path) * 10**-6, time_tot)
os.remove(path)
else:
time_start = time.time()
with modules[compression].open(path, 'wb') as fout:
pickle.dump(data[key], fout)
time_tot = time.time() - time_start
sizes[key][labels[compressions.index(compression)]] = (
os.path.getsize(path) * 10**-6,
time_tot,
)
os.remove(path)
f, ax_size = plt.subplots()
ax_time = ax_size.twinx()
x_ticks = labels
x = np.arange(len(x_ticks))
y_size = {}
y_time = {}
for key in data:
y_size[key] = [sizes[key][x_ticks[i]][0] for i in x]
y_time[key] = [sizes[key][x_ticks[i]][1] for i in x]
width = .2
viridis = plt.cm.viridis
p1 = ax_size.bar(x - width, y_size['random'], width, color = viridis(0))
p2 = ax_size.bar(x, y_size['semi-random'], width, color = viridis(.45))
p3 = ax_size.bar(x + width, y_size['not-random'], width, color = viridis(.9))
p4 = ax_time.bar(x - width, y_time['random'], .02, color='red')
ax_time.bar(x, y_time['semi-random'], .02, color='red')
ax_time.bar(x + width, y_time['not-random'], .02, color='red')
ax_size.legend(
(p1, p2, p3, p4),
('random', 'semi-random', 'not-random', 'saving time'),
loc='upper center',
bbox_to_anchor=(.5, -.1),
ncol=4,
)
ax_size.set_xticks(x)
ax_size.set_xticklabels(x_ticks)
f.suptitle('Pickle Compression Comparison')
ax_size.set_ylabel('Size [MB]')
ax_time.set_ylabel('Time [s]')
f.savefig('sizes.pdf', bbox_inches='tight')

np.load不应该使用内存映射方式加载文件。 - Fred Foonumpy.savez保存的多个数组),默认情况下是“懒加载”这些数组。它并没有将它们映射到内存,但在索引NpzFile对象之前不会加载它们。(这就是OP所指的延迟时间)。load方法的文档跳过了这一点,因此有点误导性... - Joe Kington