这是一个呼吁社区的行动,看是否有人有想法改进这个MSD计算实现的速度。这个实现主要基于这篇博客文章:http://damcb.com/mean-square-disp.html。
目前,当前的实现对于一个由5,000个点组成的二维轨迹需要大约9秒的时间。如果您需要计算许多轨迹,那么这显然太慢了...
我没有尝试并行化它(使用或),但我觉得创建新进程对于这种算法来说会太重了。
以下是代码:
输出结果为:
给出这个:
有什么想法吗?
目前,当前的实现对于一个由5,000个点组成的二维轨迹需要大约9秒的时间。如果您需要计算许多轨迹,那么这显然太慢了...
我没有尝试并行化它(使用或),但我觉得创建新进程对于这种算法来说会太重了。
以下是代码:
import os
import matplotlib
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
# Parameters
N = 5000
max_time = 100
dt = max_time / N
# Generate 2D brownian motion
t = np.linspace(0, max_time, N)
xy = np.cumsum(np.random.choice([-1, 0, 1], size=(N, 2)), axis=0)
traj = pd.DataFrame({'t': t, 'x': xy[:,0], 'y': xy[:,1]})
print(traj.head())
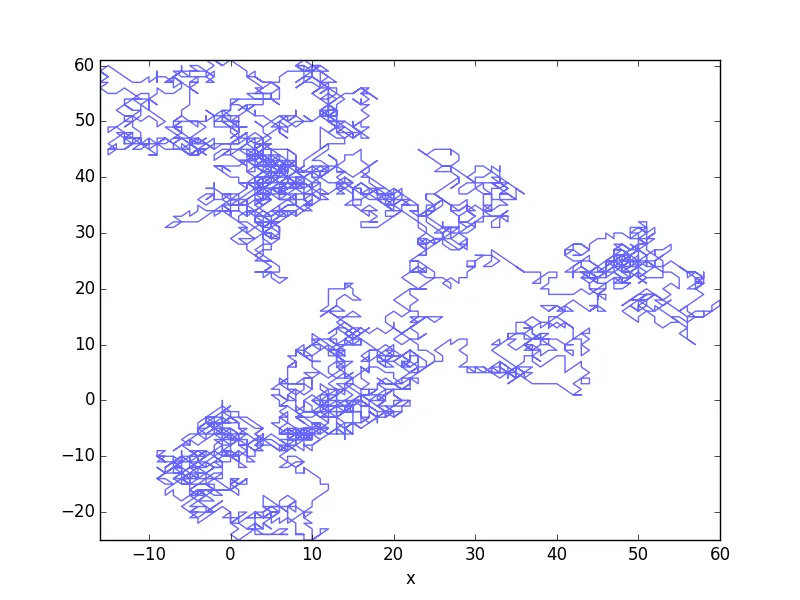
# Draw motion
ax = traj.plot(x='x', y='y', alpha=0.6, legend=False)
# Set limits
ax.set_xlim(traj['x'].min(), traj['x'].max())
ax.set_ylim(traj['y'].min(), traj['y'].max())
输出为:
t x y
0 0.000000 -1 -1
1 0.020004 -1 0
2 0.040008 -1 -1
3 0.060012 -2 -2
4 0.080016 -2 -2
def compute_msd(trajectory, t_step, coords=['x', 'y']):
tau = trajectory['t'].copy()
shifts = np.floor(tau / t_step).astype(np.int)
msds = np.zeros(shifts.size)
msds_std = np.zeros(shifts.size)
for i, shift in enumerate(shifts):
diffs = trajectory[coords] - trajectory[coords].shift(-shift)
sqdist = np.square(diffs).sum(axis=1)
msds[i] = sqdist.mean()
msds_std[i] = sqdist.std()
msds = pd.DataFrame({'msds': msds, 'tau': tau, 'msds_std': msds_std})
return msds
# Compute MSD
msd = compute_msd(traj, t_step=dt, coords=['x', 'y'])
print(msd.head())
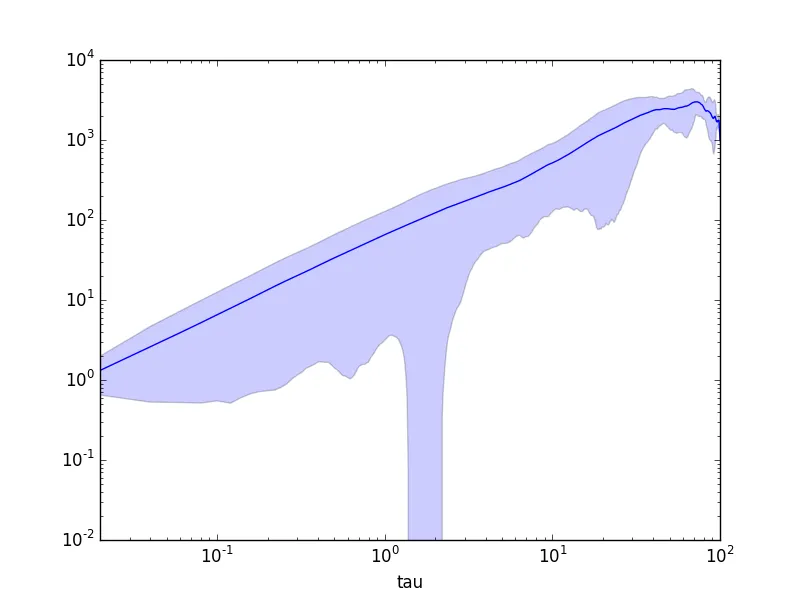
# Plot MSD
ax = msd.plot(x="tau", y="msds", logx=True, logy=True, legend=False)
ax.fill_between(msd['tau'], msd['msds'] - msd['msds_std'], msd['msds'] + msd['msds_std'], alpha=0.2)
输出结果为:
msds msds_std tau
0 0.000000 0.000000 0.000000
1 1.316463 0.668169 0.020004
2 2.607243 2.078604 0.040008
3 3.891935 3.368651 0.060012
4 5.200761 4.685497 0.080016
还有一些性能分析:
%timeit msd = compute_msd(traj, t_step=dt, coords=['x', 'y'])
给出这个:
1 loops, best of 3: 8.53 s per loop
有什么想法吗?
compute_msd的第二行中,floor函数在尝试转换为int时抛出异常。(numpy 1.9.2,Py2.7.10,OSX)还有其他人遇到这个问题吗? - rlldt = max_time / N中使用真正的除法,所以它不能在Python 2.7上工作。 - user2379410