我有一组大量的三维向量。 我需要根据欧几里得距离将它们聚类,使得任何特定簇中的所有向量彼此之间的欧几里德距离小于阈值“T”。
我不知道存在多少个簇。最后可能存在单独的向量,因为它与空间中的任何向量的欧几里德距离都不小于“T”,而不属于任何簇。
应该使用哪些现有算法/方法?
我有一组大量的三维向量。 我需要根据欧几里得距离将它们聚类,使得任何特定簇中的所有向量彼此之间的欧几里德距离小于阈值“T”。
我不知道存在多少个簇。最后可能存在单独的向量,因为它与空间中的任何向量的欧几里德距离都不小于“T”,而不属于任何簇。
应该使用哪些现有算法/方法?
import matplotlib.pyplot as plt
import numpy
import scipy.cluster.hierarchy as hcluster
# generate 3 clusters of each around 100 points and one orphan point
N=100
data = numpy.random.randn(3*N,2)
data[:N] += 5
data[-N:] += 10
data[-1:] -= 20
# clustering
thresh = 1.5
clusters = hcluster.fclusterdata(data, thresh, criterion="distance")
# plotting
plt.scatter(*numpy.transpose(data), c=clusters)
plt.axis("equal")
title = "threshold: %f, number of clusters: %d" % (thresh, len(set(clusters)))
plt.title(title)
plt.show()
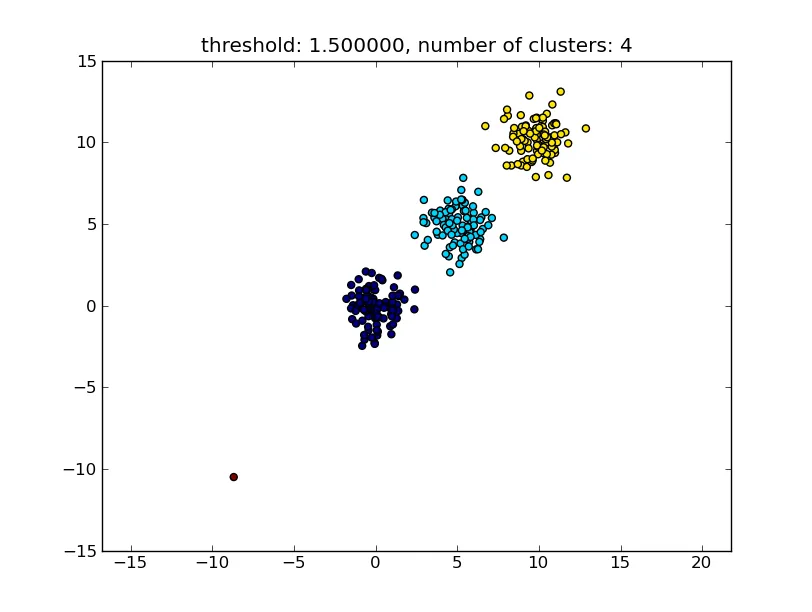 作为参数给定的阈值是一个距离值,基于此决定是否将点/簇合并到另一个簇中。也可以指定使用的距离度量。
作为参数给定的阈值是一个距离值,基于此决定是否将点/簇合并到另一个簇中。也可以指定使用的距离度量。还有其他算法可以更好地处理大量数据点的情况。正如其他答案/评论所建议的那样,您可能还想看一下DBSCAN算法:
如果您想了解更多关于这些聚类算法的信息,还可以查看Python scikit-learn库的演示页面:
从那个地方复制的图像:
正如您所见,每个算法都对需要考虑的簇的数量和形状做出了一些假设。无论是算法强加的隐含假设还是参数化指定的显式假设。
moooeeeep的回答建议使用分层聚类。我想进一步说明如何选择聚类的阈值。
一种方法是基于不同的阈值t1,t2,t3等计算聚类,然后计算聚类"质量"的指标。前提是具有最佳聚类数的聚类的质量指标将具有最大值。
我过去使用的一个好质量指标示例是Calinski-Harabasz。简而言之:计算平均簇间距离并将其除以簇内距离。最优的聚类分配将具有相互之间分离度最大的群集,并且群集最为"紧密"。
顺便说一下,您不必使用分层聚类。您还可以使用像k-means这样的东西,为每个k预先计算,然后选择具有最高Calinski-Harabasz得分的k。
如果您需要更多参考资料,请告诉我,我会在硬盘上搜索一些论文。
了解一下DBSCAN算法。它基于向量的局部密度进行聚类,即向量之间的距离不能超过某个ε值,并且可以自动确定聚类数量。此外,它还考虑了异常值,即那些没有足够数量的ε邻居点的点不会被归为任何一个簇中。维基百科页面链接了一些实现。
我想通过使用层次聚类来补充moooeeeep的答案。 尽管选择阈值相当“随意”,但这个解决方案对我很有用。 参考其他来源并进行自己的测试后,我得到了更好的方法,可以通过树状图轻松选择阈值:
from scipy.cluster import hierarchy
from scipy.spatial.distance import pdist
import matplotlib.pyplot as plt
ori_array = ["Your_list_here"]
ward_array = hierarchy.ward(pdist(ori_array))
dendrogram = hierarchy.dendrogram(hierarchy.linkage(ori_array, method = "ward"))
plt.title('Dendrogram')
plt.xlabel('Customers')
plt.ylabel('Euclidean distances')
plt.show()
threshold = 1
clusters_list = hierarchy.fcluster(ward_array, threshold, criterion="distance")
print("Clustering list: {}".format(clusters_list))
我需要一种方法来对OCR输出的行进行“模糊排序”,当输出有时是无序的,但在块内,行通常是有序的。在这种情况下,要排序的项目是描述位置'x','y'和大小'w','h'的字典。一般的聚类算法似乎过于复杂,而且我需要在排序期间保持项目的顺序。在这里,我可以将容差tol设置为大约1/4的行间距,并使用字段'y'调用它。
def fuzzy_lod_sort(lod, field, tol):
# fuzzy sort lod into bins within +/- tol
# maintain original order.
# first determine the bins.
val_list = [d[field] for d in lod]
vals_sorted = sorted(val_list)
bins_lol = []
i = 0
for j, v in enumerate(vals_sorted):
if not j:
bins_lol.append([v])
continue
cur_bin_avg = statistics.mean(bins_lol[i])
if abs(cur_bin_avg - v) <= tol:
bins_lol[i].append(v)
continue
i += 1
bins_lol.append([v])
# now sort into the bins, maintaining the original order.
# the bins will be the center of the range of 'y'.
bins = [statistics.mean(binlist) for binlist in bins_lol]
# initialize the list of bins
lolod = []
for _ in range(len(bins)):
lolod.append([])
for d in lod:
bin_idx = closest_bin_idx(bins, d[field])
lolod[bin_idx].append(d)
# now join the bins.
result_lod = []
for lod in lolod:
result_lod.extend(lod)
return result_lod
def closest_bin(bins, val):
return min(bins, key=lambda bin:abs(bin - val))
def closest_bin_idx(bins, val):
return bins.index(closest_bin(bins, val))
也许有一些方式使用内置排序进行模糊排序,这可能是一维问题聚类选项的替代方案。
DBSCAN。 - Has QUIT--Anony-MousseDBSCAN带有示例用法:https://scikit-learn.org/stable/modules/generated/sklearn.cluster.DBSCAN.html#sklearn.cluster.DBSCAN - Jean Monet