我刚刚完成了Python中的贝叶斯分析一书,作者是Osvaldo Martin(这是一本非常好的书,可以帮助您理解贝叶斯概念和一些花式numpy索引)。
我真的想扩展我的理解,以便于使用贝叶斯混合模型对样本进行无监督聚类。我所有的谷歌搜索都导向了Austin Rochford的教程,它真的很有启发性。我理解正在发生的事情,但我不清楚如何将其适应于聚类(特别是在使用多个属性进行聚类分配时,但这是一个不同的话题)。
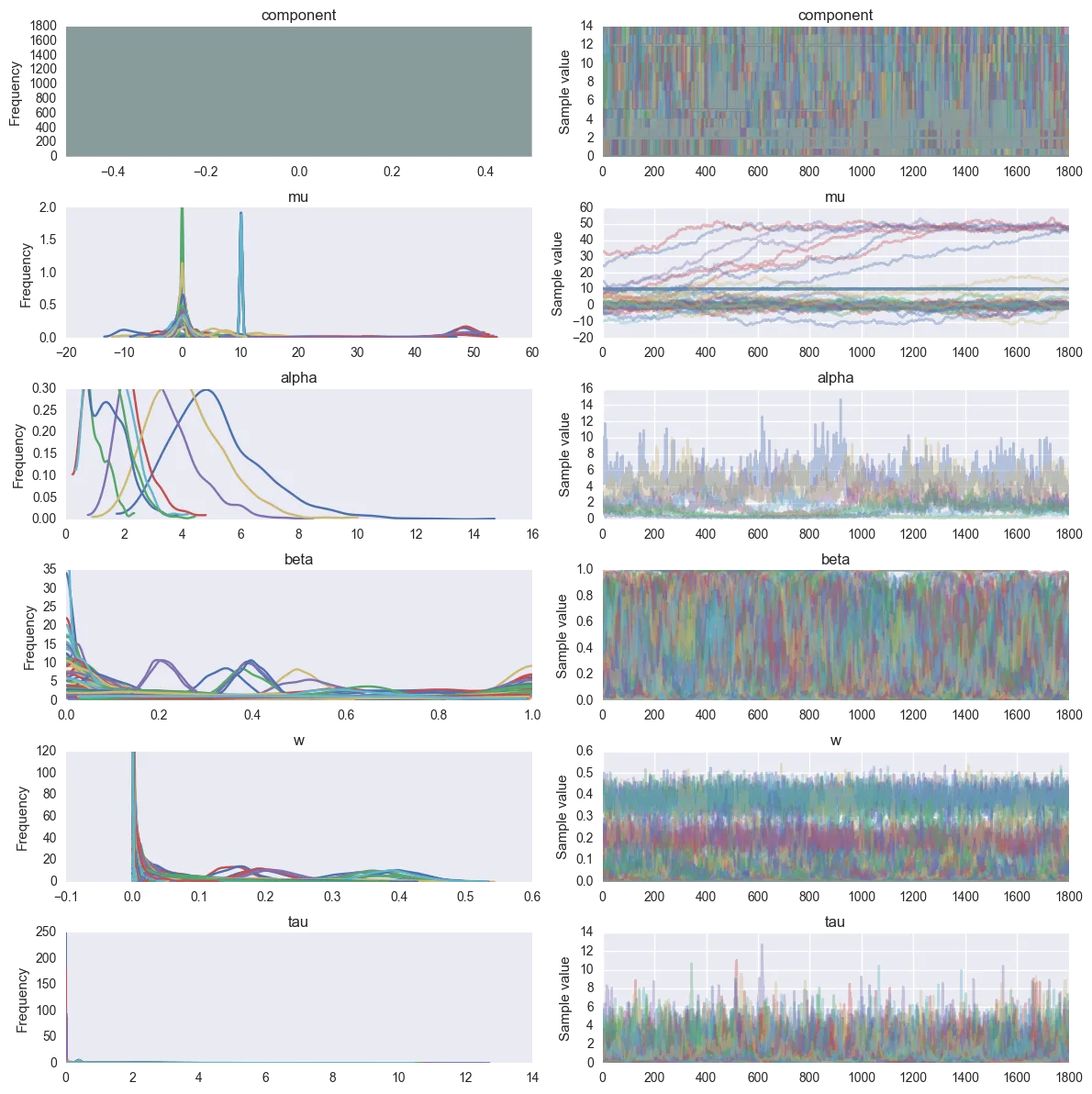
我知道如何为Dirichlet分布分配先验概率,但我不知道如何在PyMC3中获取聚类。看起来大多数mus会收敛到质心(即我从中抽样的分布的均值),但它们仍然是独立的组件。我考虑过为权重(模型中的w)制定截止值,但这似乎并不像我想象的那样有效,因为多个具有略微不同平均参数mu的组件正在收敛。
我该如何从这个PyMC3模型中提取聚类(质心)?我将其设置为最多有15个组件,并希望收敛到3个。mus似乎位于正确位置,但权重混乱了,因为它们分布在其他聚类之间,所以我不能使用权重阈值(除非我合并它们,但我认为这不是正常做法)。
我真的想扩展我的理解,以便于使用贝叶斯混合模型对样本进行无监督聚类。我所有的谷歌搜索都导向了Austin Rochford的教程,它真的很有启发性。我理解正在发生的事情,但我不清楚如何将其适应于聚类(特别是在使用多个属性进行聚类分配时,但这是一个不同的话题)。
我知道如何为Dirichlet分布分配先验概率,但我不知道如何在PyMC3中获取聚类。看起来大多数mus会收敛到质心(即我从中抽样的分布的均值),但它们仍然是独立的组件。我考虑过为权重(模型中的w)制定截止值,但这似乎并不像我想象的那样有效,因为多个具有略微不同平均参数mu的组件正在收敛。
我该如何从这个PyMC3模型中提取聚类(质心)?我将其设置为最多有15个组件,并希望收敛到3个。mus似乎位于正确位置,但权重混乱了,因为它们分布在其他聚类之间,所以我不能使用权重阈值(除非我合并它们,但我认为这不是正常做法)。
import pymc3 as pm
import numpy as np
import matplotlib.pyplot as plt
import multiprocessing
import seaborn as sns
import pandas as pd
import theano.tensor as tt
%matplotlib inline
# Clip at 15 components
K = 15
# Create mixture population
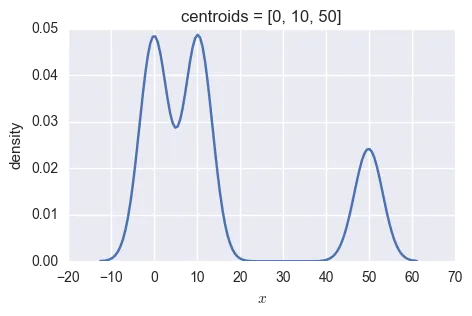
centroids = [0, 10, 50]
weights = [(2/5),(2/5),(1/5)]
mix_3 = np.concatenate([np.random.normal(loc=centroids[0], size=int(150*weights[0])), # 60 samples
np.random.normal(loc=centroids[1], size=int(150*weights[1])), # 60 samples
np.random.normal(loc=centroids[2], size=int(150*weights[2]))])# 30 samples
n = mix_3.size
# Create and fit model
with pm.Model() as Mod_dir:
alpha = pm.Gamma('alpha', 1., 1.)
beta = pm.Beta('beta', 1., alpha, shape=K)
w = pm.Deterministic('w', beta * tt.concatenate([[1], tt.extra_ops.cumprod(1 - beta)[:-1]]))
component = pm.Categorical('component', w, shape=n)
tau = pm.Gamma("tau", 1.0, 1.0, shape=K)
mu = pm.Normal('mu', 0, tau=tau, shape=K)
obs = pm.Normal('obs',
mu[component],
tau=tau[component],
observed=mix_3)
step1 = pm.Metropolis(vars=[alpha, beta, w, tau, mu, obs])
# step2 = pm.CategoricalGibbsMetropolis(vars=[component])
step2 = pm.ElemwiseCategorical([component], np.arange(K)) # Much, much faster than the above
tr = pm.sample(1e4, [step1, step2], njobs=multiprocessing.cpu_count())
#burn-in = 1000, thin by grabbing every 5th idx
pm.traceplot(tr[1e3::5])
以下是类似的问题
https://stats.stackexchange.com/questions/120209/pymc3-dirichlet-distribution 用于回归而不是聚类
https://stats.stackexchange.com/questions/108251/image-clustering-and-dirichlet-process DP过程的理论
Dirichlet process in PyMC 3 将我指向了Austin Rochford的上面的教程