我认为使用graphviz的
twopi布局没有单个图形的方法来实现这一点。Twopi通常可以很好地设置每个子图的根节点,因为如
文档中所述,它会随机选择距离叶节点最远的节点之一作为根节点,所以在只有一个根节点的情况下,这应该导致预期的拓扑结构排列。但是,如果不是这种情况,并且您想要为每个子图手动设置根节点,则我的方法是通过迭代连接组分子图中的图形,并将每个组件绘制到subplot图中的单独轴中,为每个创建自定义
graphviz_layout。
以下是使用以下示例图形完成此操作的方法:
from matplotlib import pyplot as plt
import pygraphviz
from networkx.drawing.nx_agraph import graphviz_layout
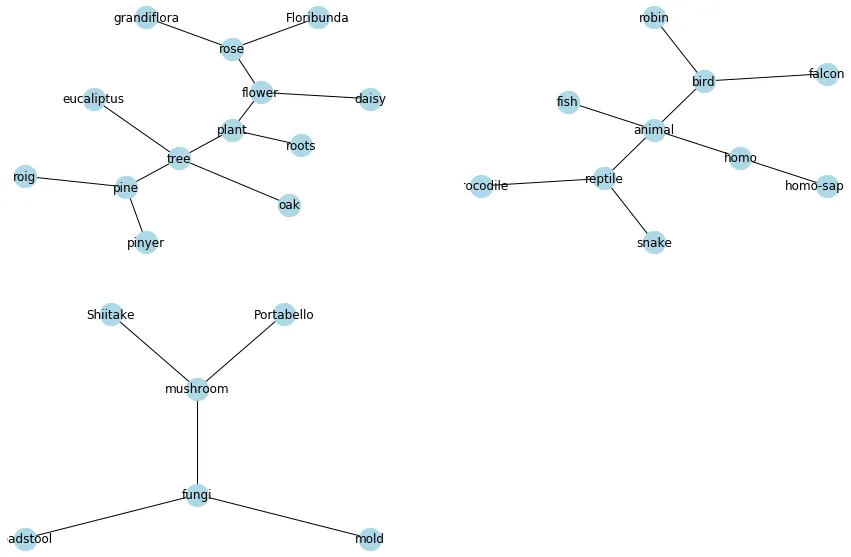
result_set = {('plant','tree'), ('tree','oak'), ('flower', 'rose'), ('flower','daisy'), ('plant','flower'), ('tree','pine'), ('plant','roots'), ('animal','fish'),('animal','bird'), ('bird','robin'), ('bird','falcon'), ('animal', 'homo'),('homo','homo-sapiens'), ('animal','reptile'), ('reptile','snake'),('fungi','mushroom'), ('fungi','mold'), ('fungi','toadstool'),('reptile','crocodile'), ('mushroom','Portabello'), ('mushroom','Shiitake'),('pine','roig'),('pine','pinyer'), ('tree','eucaliptus'),('rose','Floribunda'),('rose','grandiflora')}
G=nx.from_edgelist(result_set, create_using=nx.DiGraph)
为了遍历现有的子图,如果当前图不是无向图,则必须将其复制为无向图,并使用
nx.connected_component_subgraphs 创建子图列表:
UG = G.to_undirected()
subgraphs = list(nx.connected_component_subgraphs(UG))
假设我们知道我们希望不同组件的根节点是节点
'plant',
'animal'和
'mushroom',我们现在可以创建一组子图,遍历相应的坐标轴、子图对象和根列表(确保它们按照相同的顺序),为每个子图创建一个新布局,并设置相应的根节点。
n_cols = 2
roots = ['plant','animal','mushroom']
fig, axes = plt.subplots(nrows=int(np.ceil(len(subgraphs)/n_cols)),
ncols=n_cols,
figsize=(15,10))
plt.box(False)
for subgraph, root, ax in zip(subgraphs, roots, axes.flatten()):
pos = graphviz_layout(G, prog='twopi', args=f"-Groot={root}")
nx.draw(subgraph, pos=pos, with_labels=True,
node_color='lightblue', node_size=500, ax=ax)