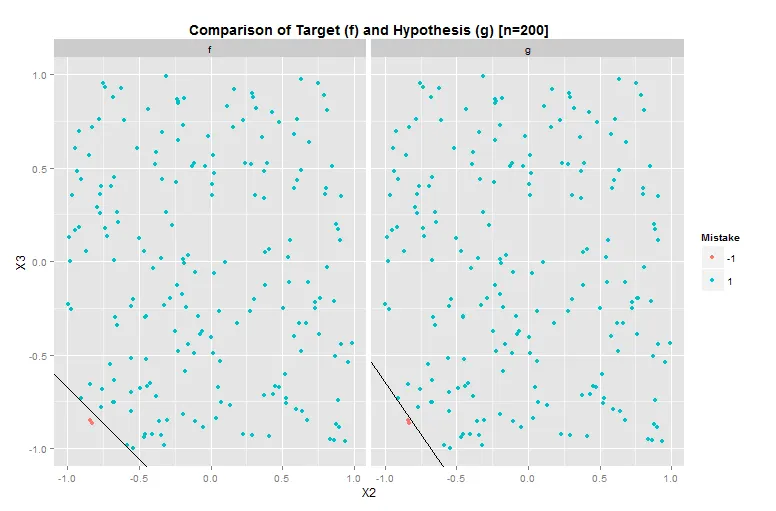
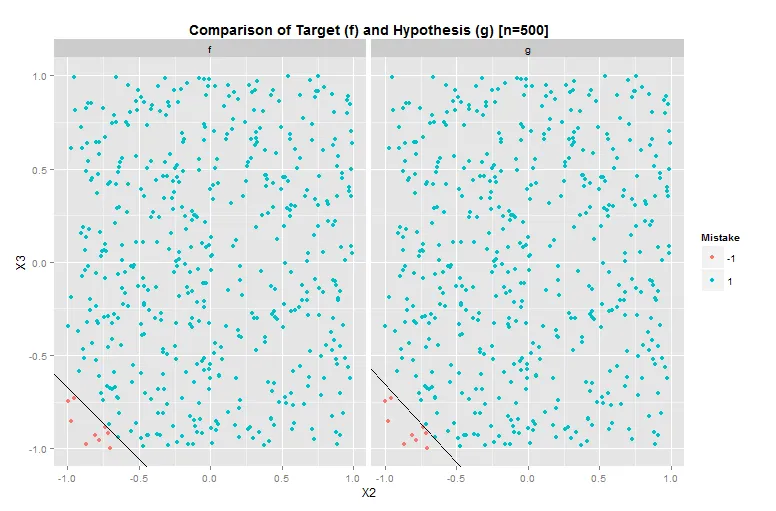
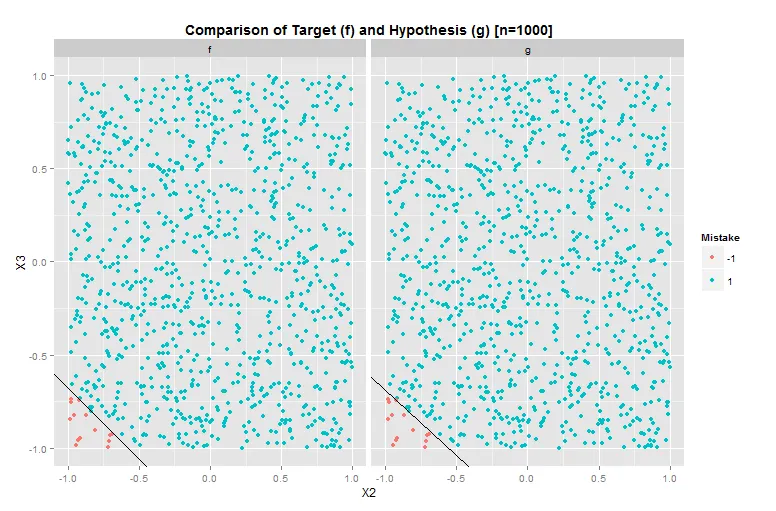
你的例子仍无法重现。看看我的代码,你会发现f和g是完全相同的。此外,你似乎正在对你没有的数据点进行线性外推(你问题的第二部分)。你有证据表明歧视应该是线性的吗?
n <- 10000
datam <- matrix(c(rep(1,n), 2*runif(n)-1, 2*runif(n)-1), n)
datam.df<-data.frame(datam)
datam.df$X1<-NULL
f <- c(1.0, 0.5320523, 0.6918301)
f.col <- ifelse(sign(datam %*% f)==1,"darkred", "darkblue")
f.fun<-sign(datam %*% f)
perceptron = function(datam, ylist) {
w <- c(1,0,0)
made.mistake = TRUE
while (made.mistake) {
made.mistake=FALSE
for (i in 1:n) {
if (ylist[i] != sign(t(w) %*% datam[i,])) {
w <- w + ylist[i]*datam[i,]
made.mistake=TRUE
}
}
}
return(w=w)
}
g <- perceptron(datam, f.fun)
g.fun<-sign(datam %*% g)
绘制整体数据
plot(datam.df$X2, datam.df$X3, col=f.col, pch=".", cex=2)
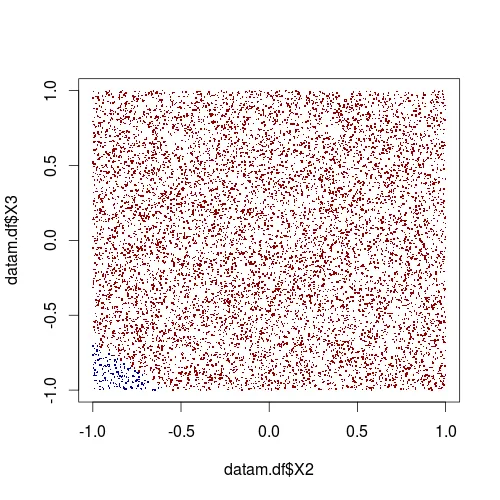
针对g和f函数存在问题,我会分别制作独立的图表。在你解决这个问题之后,你可以将所有东西放在一个图表中。你还可以查看并选择是否需要阴影。如果没有证据表明分类是线性的,使用 chull() 标记你所拥有的数据可能更加明智。
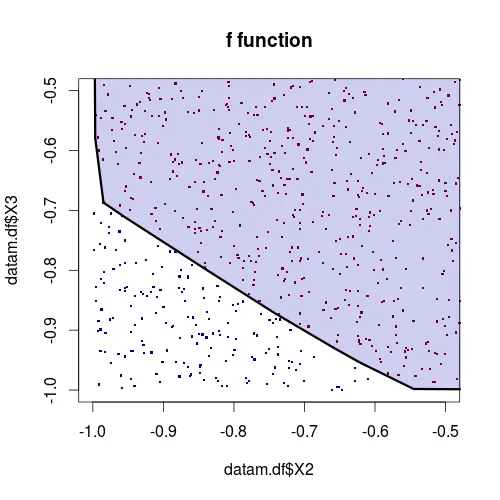
对于f函数
plot(datam.df$X2, datam.df$X3, col=f.col, pch=".", xlim=c(-1,-0.5), ylim=c(-1,-.5), cex=3, main="f function")
datam.df.f<-datam.df[f.fun==1,]
ch.f<-chull(datam.df.f$X2, datam.df.f$X3 )
ch.f <- rbind(x = datam.df.f[ch.f, ], datam.df.f[ch.f[1], ])
polygon(ch.f, lwd=3, col=rgb(0,0,180,alpha=50, maxColorValue=255))
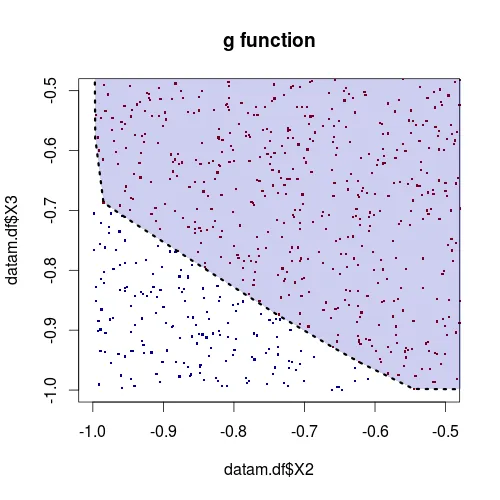
针对g函数
g.col <- ifelse(sign(datam %*% g)==1,"darkred", "darkblue")
plot(datam.df$X2, datam.df$X3, col=g.col, pch=".", xlim=c(-1,-0.5), ylim=c(-1,-.5), cex=3, main="g function")
datam.df.g<-datam.df[g.fun==1,]
ch.g<-chull(datam.df.g$X2, datam.df.g$X3 )
ch.g <- rbind(x = datam.df.g[ch.g, ], datam.df.g[ch.g[1], ])
polygon(ch.g, col=rgb(0,0,180,alpha=50, maxColorValue=255), lty=3, lwd=3)
ch.f和ch.g对象是围绕您的点的“包”坐标。您可以提取这些点来描述您的线。
注:原文已经是英文,所以无需翻译,只需要将其翻译成中文即可。
ch.f
lm.f<-lm(c(ch.f$X3[ ch.f$X2> -0.99 & ch.f$X2< -0.65 & ch.f$X3<0 ])~c(ch.f$X2[ ch.f$X2>-0.99 & ch.f$X2< -0.65 & ch.f$X3<0]))
curve(lm.f$coefficients[1]+x*lm.f$coefficients[2], from=-1., to=-0.59, lwd=5, add=T)
lm.g<-lm(c(ch.g$X3[ ch.g$X2> -0.99 & ch.g$X2< -0.65 & ch.g$X3<0 ])~c(ch.g$X2[ ch.g$X2>-0.99 & ch.g$X2< -0.65 & ch.g$X3<0]))
curve(lm.g$coefficients[1]+x*lm.g$coefficients[2], from=-1., to=-0.59, lwd=5, add=T, lty=3)
您会得到
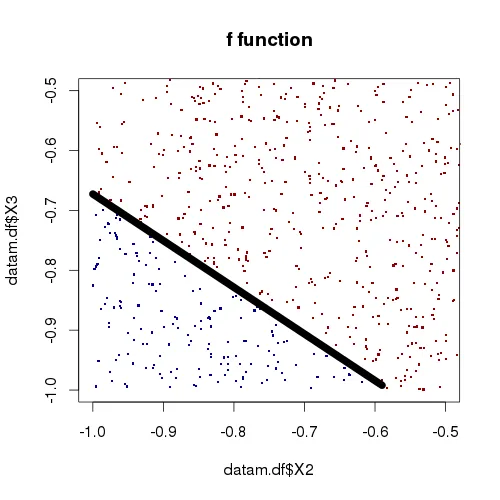
不幸的是,由于您的示例中f和g函数相同,因此您无法看到上图中的不同行。
 这个Mathematica图显示了f和g的比较(不同的数据集和f):
这个Mathematica图显示了f和g的比较(不同的数据集和f):
 这是相应的Mathematica代码。
这是相应的Mathematica代码。