我正在尝试模拟一种粒子在电斥力(或吸引力)作用下飞向另一种粒子的现象,称为卢瑟福散射。我已经成功使用for循环和Python列表模拟了一些粒子。但是,现在我想使用numpy数组。该模型将采用以下步骤:
1. 对于所有粒子: 1. 计算与所有其他粒子的径向距离 2. 计算与所有其他粒子的夹角 3. 计算x方向和y方向的净力 2. 创建每个粒子的净xForce和yForce矩阵 3. 通过a = F / mass创建加速度(也是x和y分量)矩阵 4. 更新速度矩阵 5. 更新位置矩阵
我的问题是我不知道如何在计算力的分量时使用numpy数组。 以下是我的代码,但无法运行。
1. 对于所有粒子: 1. 计算与所有其他粒子的径向距离 2. 计算与所有其他粒子的夹角 3. 计算x方向和y方向的净力 2. 创建每个粒子的净xForce和yForce矩阵 3. 通过a = F / mass创建加速度(也是x和y分量)矩阵 4. 更新速度矩阵 5. 更新位置矩阵
我的问题是我不知道如何在计算力的分量时使用numpy数组。 以下是我的代码,但无法运行。
import numpy as np
# I used this function to calculate the force while using for-loops.
def force(x1, y1, x2, x2):
angle = math.atan((y2 - y1)/(x2 - x1))
dr = ((x1-x2)**2 + (y1-y2)**2)**0.5
force = charge2 * charge2 / dr**2
xforce = math.cos(angle) * force
yforce = math.sin(angle) * force
# The direction of force depends on relative location
if x1 > x2 and y1<y2:
xforce = xforce
yforce = yforce
elif x1< x2 and y1< y2:
xforce = -1 * xforce
yforce = -1 * yforce
elif x1 > x2 and y1 > y2:
xforce = xforce
yforce = yforce
else:
xforce = -1 * xforce
yforce = -1* yforce
return xforce, yforce
def update(array):
# this for loop defeats the entire use of numpy arrays
for particle in range(len(array[0])):
# find distance of all particles pov from 1 particle
# find all x-forces and y-forces on that particle
xforce = # sum of all x-forces from all particles
yforce = # sum of all y-forces from all particles
force_arr[0, particle] = xforce
force_arr[1, particle] = yforce
return force
# begin parameters
t = 0
N = 3
masses = np.ones(N)
charges = np.ones(N)
loc_arr = np.random.rand(2, N)
speed_arr = np.random.rand(2, N)
acc_arr = np.random.rand(2, N)
force = np.random.rand(2, N)
while t < 0.5:
force_arr = update(loc_arry)
acc_arr = force_arr / masses
speed_arr += acc_array
loc_arr += speed_arr
t += dt
# plot animation
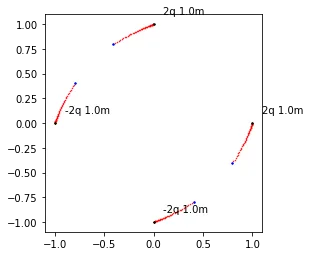
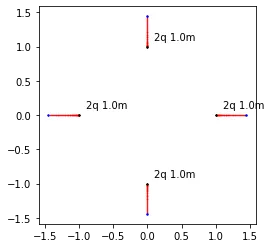
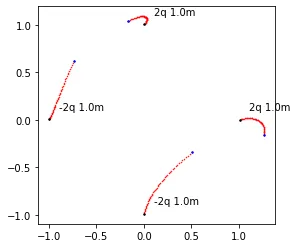
![masses[0] = 5](https://istack.dev59.com/aEcrj.webp)
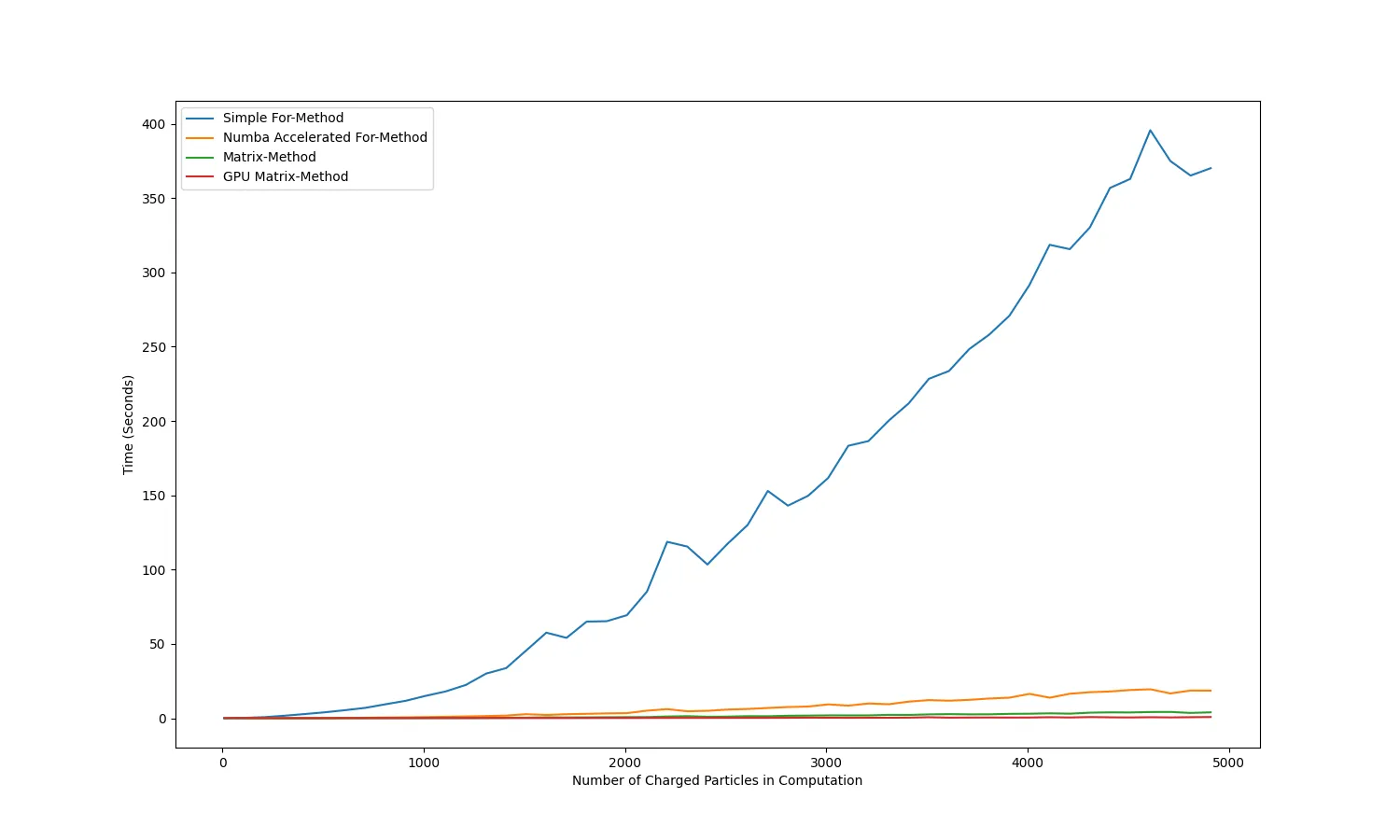
# this for loop defeats the entire use of numpy arrays表示你想避免使用循环,但是想要进行“内联”计算?也许向量化是一个好的选择:https://towardsdatascience.com/python-vectorization-5b882eeef658 - L.Clarkson