背景
我成功地复现了这种行为:
- 在Win上 - 但这不应该是一个问题,因为(我假设)在OSX上情况是相同的
- (当前)Anaconda环境下的Python和Anaconda默认环境下的Python是不同的。如果不是这种情况,答案(或部分答案)可能是不正确的。请注意,这是我要引用的唯一两个Python,它们都是Anaconda内部的(即使Anaconda“关键字”不会出现)
- 我在我的测试场景中使用了PyGraphviz(而不是PyOpenCL)
code0.py:
import sys
import os
import pprint
print(f"Python Executable: {sys.executable}")
print(f"Version {sys.version} on {sys.platform}\n")
conda_env_var = "CONDA_DEFAULT_ENV"
conda_env = os.environ[conda_env_var]
print(f"{conda_env_var}: {conda_env}\n")
sys_path = pprint.pformat(sys.path)
print(f"sys.path: {sys_path}\n")
path_var = "PATH"
env_path = pprint.pformat([item for item in os.environ[path_var].split(os.pathsep) if item.find(conda_env) > -1])
print(f"os.environ[\"{path_var}\"] (relevant): {env_path}\n")
import pygraphviz
print(pygraphviz)
Output:
(py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> python code0.py
Python Executable: E:\Install\x64\Anaconda\Anaconda\2018.12\envs\py_064_030701_test0\python.exe
Version 3.7.1 (default, Dec 10 2018, 22:54:23) [MSC v.1915 64 bit (AMD64)] on win32
CONDA_DEFAULT_ENV: py_064_030701_test0
sys.path: ['e:\\Work\\Dev\\StackOverflow\\q055251357',
'E:\\Work\\Dev\\Utils',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\python37.zip',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\DLLs',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\lib',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\lib\\site-packages']
os.environ["PATH"] (relevant): ['E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\Library\\mingw-w64\\bin',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\Library\\usr\\bin',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\Library\\bin',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\Scripts',
'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\bin']
<module 'pygraphviz' from 'E:\\Install\\x64\\Anaconda\\Anaconda\\2018.12\\envs\\py_064_030701_test0\\lib\\site-packages\\pygraphviz\\__init__.py'>
(py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> where jupyter-notebook
E:\Install\x64\Anaconda\Anaconda\2018.12\Scripts\jupyter-notebook.exe
(py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> jupyter-notebook
[I 01:16:10.345 NotebookApp] JupyterLab extension loaded from E:\Install\x64\Anaconda\Anaconda\2018.12\lib\site-packages\jupyterlab
[I 01:16:10.346 NotebookApp] JupyterLab application directory is E:\Install\x64\Anaconda\Anaconda\2018.12\share\jupyter\lab
[I 01:16:10.349 NotebookApp] Serving notebooks from local directory: e:\Work\Dev\StackOverflow\q055251357
[I 01:16:10.350 NotebookApp] The Jupyter Notebook is running at:
[I 01:16:10.352 NotebookApp] http://localhost:8888/?token=14412a6d6d0c895d059a86bcd71e10cbface4a479c5843c2
[I 01:16:10.353 NotebookApp] Use Control-C to stop this server and shut down all kernels (twice to skip confirmation).
[C 01:16:10.437 NotebookApp]
To access the notebook, open this file in a browser:
file:///C:/Users/cfati/AppData/Roaming/jupyter/runtime/nbserver-24700-open.html
Or copy and paste one of these URLs:
http://localhost:8888/?token=14412a6d6d0c895d059a86bcd71e10cbface4a479c5843c2
[I 01:17:18.569 NotebookApp] 302 GET /?token=14412a6d6d0c895d059a86bcd71e10cbface4a479c5843c2 (::1) 0.98ms
[I 01:17:25.161 NotebookApp] Creating new notebook in
[I 01:17:26.147 NotebookApp] Kernel started: 8b702b2d-97d0-40e3-bbca-42107efd1de5
[I 01:17:27.186 NotebookApp] Adapting to protocol v5.1 for kernel 8b702b2d-97d0-40e3-bbca-42107efd1de5
同样的脚本也在 Jupyter Notebook 中运行:
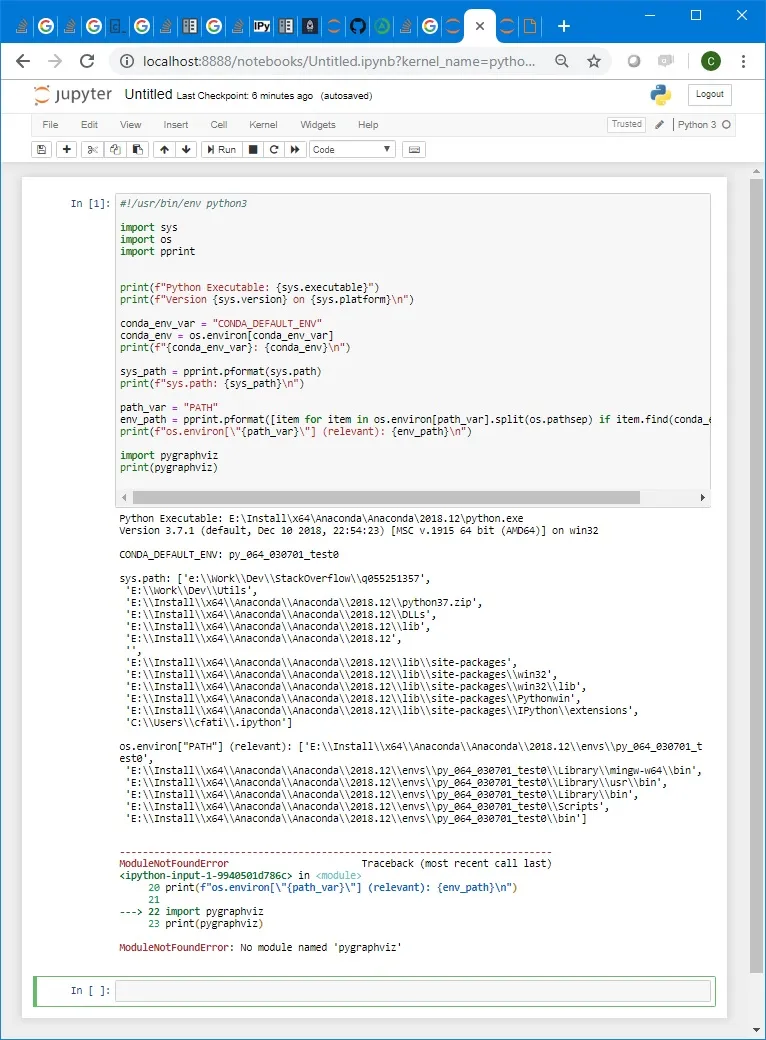
如所见,它失败了,这是因为它是由
Anaconda的默认
Python运行的(该软件包未安装)。看了一下发现,
jupyter-notebook可执行文件启动了(
Anaconda的默认)
Python在同一目录下的
jupyter-notebook-script.py上。
可能的解决方案:
1. 在主要的Python中安装缺失的软件包
这是我首先想到的:安装PyGraphviz(和所有其他所需的软件包)。 没有尝试过,但应该可以工作。我之所以没有尝试,是因为我反对用软件包污染主要的Python。但是,由于它已经包含了大量的site-packages,事情是值得商榷的。
2. 注册当前环境Python为内核
我试图让
jupyter-notebook 使用当前环境的 Python 安装,使用它的配置或修改
%CONDA_PYTHON_EXE%,但没有成功(请注意这是我第一次使用 Jupyter)。无论如何,在进行一些调查后,我意识到
jupyter-notebook可执行文件启动了安装在其中的 Jupyter 的 Python。这是一种常见的技术,通过将 Python 路径硬编码到可执行文件中来实现(尽管奇怪的是,用十六进制编辑器查看时找不到它)。
在搜索过程中,我发现
[SO]: Changing Python Executable (@Matt's answer),然后跳转到
[ReadTheDocs.IPython]: Installing the IPython kernel并尝试了一下。
py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> where pip
E:\Install\x64\Anaconda\Anaconda\2018.12\envs\py_064_030701_test0\Scripts\pip.exe
E:\Install\x64\Anaconda\Anaconda\2018.12\Scripts\pip.exe
(py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> pip freeze
certifi==2019.3.9
pygraphviz==1.5
wincertstore==0.2
(py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> pip install ipykernel
Collecting ipykernel
...
# Some pip useless output
...
Installing collected packages: tornado, colorama, six, ipython-genutils, decorator, traitlets, backcall, pygments, pickleshare, wcwidth, prompt-toolkit, parso, jedi, ipython, jupyter-core, python-dateutil, pyzmq, jupyter-client, ipykernel
Successfully installed backcall-0.1.0 colorama-0.4.1 decorator-4.4.0 ipykernel-5.1.0 ipython-7.4.0 ipython-genutils-0.2.0 jedi-0.13.3 jupyter-client-5.2.4 jupyter-core-4.4.0 parso-0.3.4 pickleshare-0.7.5 prompt-toolkit-2.0.9 pygments-2.3.1 python-dateutil-2.8.0 pyzmq-18.0.1 six-1.12.0 tornado-6.0.2 traitlets-4.3.2 wcwidth-0.1.7
(py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> where python
E:\Install\x64\Anaconda\Anaconda\2018.12\envs\py_064_030701_test0\python.exe
E:\Install\x64\Anaconda\Anaconda\2018.12\python.exe
(py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> python -m ipykernel install --name %CONDA_DEFAULT_ENV%
Installed kernelspec py_064_030701_test0 in C:\ProgramData\jupyter\kernels\py_064_030701_test0
启动后,选择新创建的内核(如下图所示),一切都很顺利。
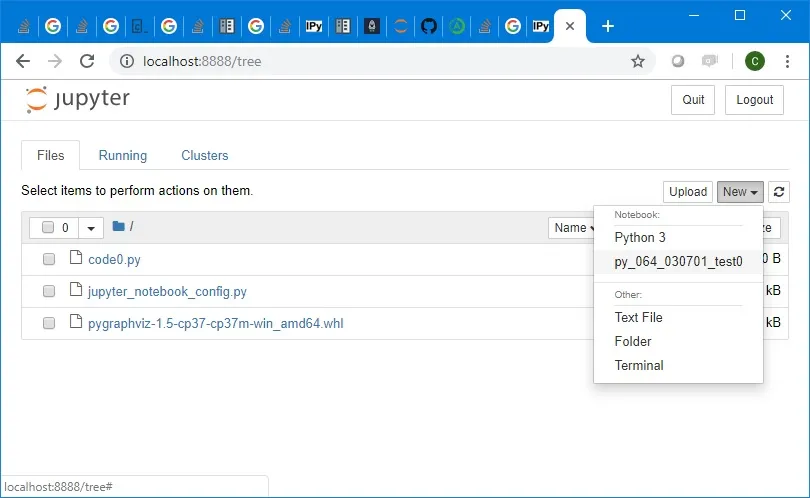
这基本上就是@AndrásNagy在他的答案中解释的内容。
虽然这是我一开始的首选,但使用当前环境下的Python将其元数据编写到一个位置,以便主要的Python (以及其他不一定在Anaconda内的Python)可以从中读取,对我来说似乎并不那么直接(尽管这可能是推荐的方法)。
3. 在当前环境的Python中安装Jupyter
我一开始也考虑了这个方法,但由于之前的方法,我没有立即开始。我认为Jupyter有很多依赖项(这是真的),但IPyKernel也有很多依赖项。然而,现在我认为这是最简单的方法。
(py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> pip install jupyter
Collecting jupyter
...
# Some pip useless output
...
Installing collected packages: qtconsole, testpath, defusedxml, entrypoints, webencodings, bleach, mistune, MarkupSafe, jinja2, pandocfilters, attrs, pyrsistent, jsonschema, nbformat, nbconvert, Send2Trash, prometheus-client, pywinpty, terminado, notebook, widgetsnbextension, ipywidgets, jupyter-console, jupyter
Successfully installed MarkupSafe-1.1.1 Send2Trash-1.5.0 attrs-19.1.0 bleach-3.1.0 defusedxml-0.5.0 entrypoints-0.3 ipywidgets-7.4.2 jinja2-2.10 jsonschema-3.0.1 jupyter-1.0.0 jupyter-console-6.0.0 mistune-0.8.4 nbconvert-5.4.1 nbformat-4.4.0 notebook-5.7.8 pandocfilters-1.4.2 prometheus-client-0.6.0 pyrsistent-0.14.11 pywinpty-0.5.5 qtconsole-4.4.3 terminado-0.8.2 testpath-0.4.2 webencodings-0.5.1 widgetsnbextension-3.4.2
(py_064_030701_test0) [cfati@CFATI-5510-0:e:\Work\Dev\StackOverflow\q055251357]> where jupyter-notebook
E:\Install\x64\Anaconda\Anaconda\2018.12\envs\py_064_030701_test0\Scripts\jupyter-notebook.exe
E:\Install\x64\Anaconda\Anaconda\2018.12\Scripts\jupyter-notebook.exe
毋庸置疑,启动
jupyter-notebook (
注意它是不同的可执行文件)就解决了问题(因为安装
Jupyter 也将
Python 安装作为一个内核注册了)。


print(cl)并粘贴输出? - CristiFati