我创建了一个箱线图,展示了一些物种
(对于密度,我计算了每个物种的比例,因此从10到100。它在列
$spe的分散距离$dist,我希望箱子的宽度与这些物种再生密度成比例。我使用了"varwidth"和权重美学,如下所示,但仍然不正确,因为它仅与观测数量成比例,而不是与再生密度成比例...(对于密度,我计算了每个物种的比例,因此从10到100。它在列
data_dist2$prop2中给出。)p <- ggplot(data_dist2, aes(x = reorder(spe, prop2), y = dist)) +
coord_flip() +
geom_boxplot(varwidth = TRUE, alpha=0.3, aes(weight=data_dist2$prop2), fill='grey10')
请问您是否有任何想法如何使箱线图完全与我的 prop2 列成比例?
示例:
structure(list(spe = structure(c(1L, 1L, 1L, 1L, 1L, 1L, 1L,
1L, 1L, 1L, 1L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L,
3L, 3L, 3L, 3L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L,
2L), .Label = c("Abies concolor", "Picea abies", "Sequoia semp."
), class = "factor"), dist = c(0, 0, 3, 3, 4, 4, 25, 46, 59,
113, 113, 9, 12, 12, 12, 15, 22, 22, 22, 22, 35, 35, 36, 49,
85, 85, 90, 5, 5, 1, 1, 8, 13, 48, 48, 52, 52, 52, 65, 89), prop2 = c(92.17,
92.17, 92.17, 92.17, 92.17, 92.17, 92.17, 92.17, 92.17, 92.17,
92.17, 10.9, 10.9, 10.9, 10.9, 10.9, 10.9, 10.9, 10.9, 10.9,
10.9, 10.9, 10.9, 10.9, 10.9, 10.9, 10.9, 100, 100, 100, 100,
100, 100, 100, 100, 100, 100, 100, 100, 100)), row.names = c(NA,
-40L), class = "data.frame")
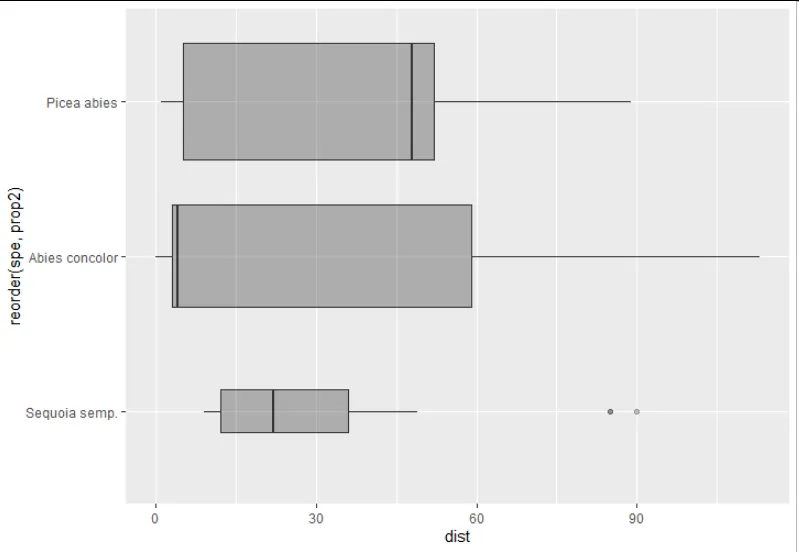
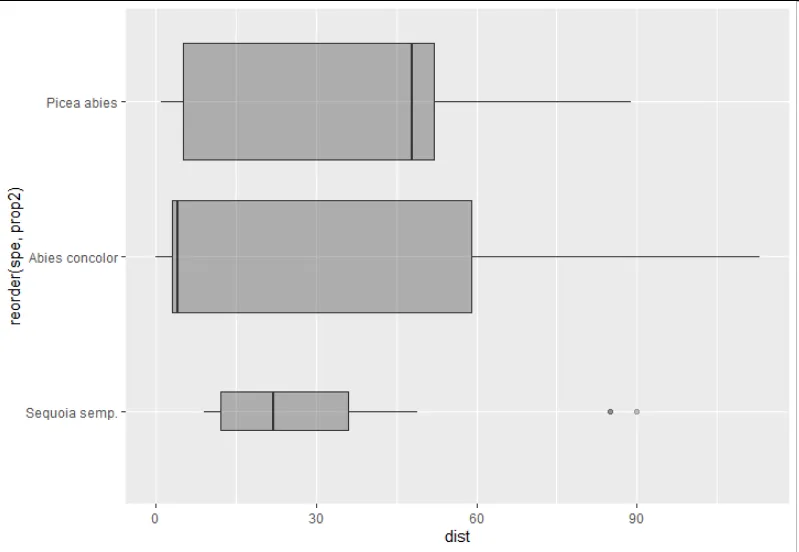
prop2的范围在10到100之间,您可以设置aes(width = I(prop2 / 100))。这将把宽度设置为从0.1到1。 - Drumygeom_boxplot接受width作为美学参数。我的错。解决方案比那复杂一点。请看下面的答案。 - Drumy