您的问题很难以最优方式解决(即找到最少的组数)。正如评论中所指出的,您的方法是依赖于顺序的:
[0, 0.006, 0.004]将产生两个组(
[0, 0.005]),而
[0, 0.004, 0.006]将产生一个单一的组(
[0.0033..])。此外,这是一种贪心聚合分组方法,它削减了许多可能的组,通常包括最优组。
以下是使用聚合聚类的一种方法。对于n个点,它的时间复杂度介于
O(n log n)和
O(n^2)之间:在我的机器上,1K个点大约需要61毫秒,而5K个点需要3.2秒。它还需要稍微更改定义:我们通过“中心”(边界框的中心)来表示组,而不是平均质心。
我们使用
linkage='complete'链接类型(因此群集的总直径是决定性指标),并将最大距离(即直径)设置为您的“公差”的两倍。
示例
from sklearn.cluster import AgglomerativeClustering
def quantize(df, tolerance=0.005):
model = AgglomerativeClustering(distance_threshold=2 * tolerance, linkage='complete',
n_clusters=None).fit(df)
df = df.assign(
group=model.labels_,
center=df.groupby(model.labels_).transform(lambda v: (v.max() + v.min()) / 2),
)
return df
根据您的数据,处理时间为4.4毫秒,并得到以下df结果:
>>> quantize(df[['Result']], tolerance=0.005)
Result group center
0 0.001 0 0.0020
1 0.000 0 0.0020
2 -0.001 0 0.0020
3 0.005 0 0.0020
4 0.002 0 0.0020
5 0.003 0 0.0020
6 0.004 0 0.0020
7 0.001 0 0.0020
8 3.400 2 3.4025
9 3.401 2 3.4025
10 3.405 2 3.4025
11 3.402 2 3.4025
12 0.003 0 0.0020
13 0.004 0 0.0020
14 0.001 0 0.0020
15 4.670 1 4.6725
16 -0.001 0 0.0020
17 4.675 1 4.6725
18 4.672 1 4.6725
19 0.003 0 0.0020
20 3.404 2 3.4025
可视化
您可以使用scipy.cluster.hierarchy.dendrogram来可视化相应的树状图:
from scipy.cluster.hierarchy import dendrogram, linkage
from scipy.spatial.distance import pdist
Z = linkage(pdist(df[['Result']]), 'complete')
dn = dendrogram(Z)
plt.axhline(2 * tolerance, c='k')
plt.ylim(0, 2.1 * tolerance)
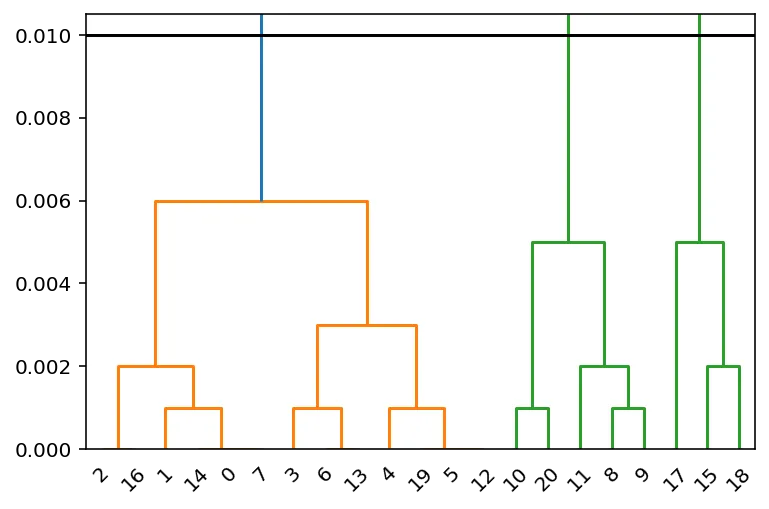
在 2 * 容忍度 下方有三个聚类。
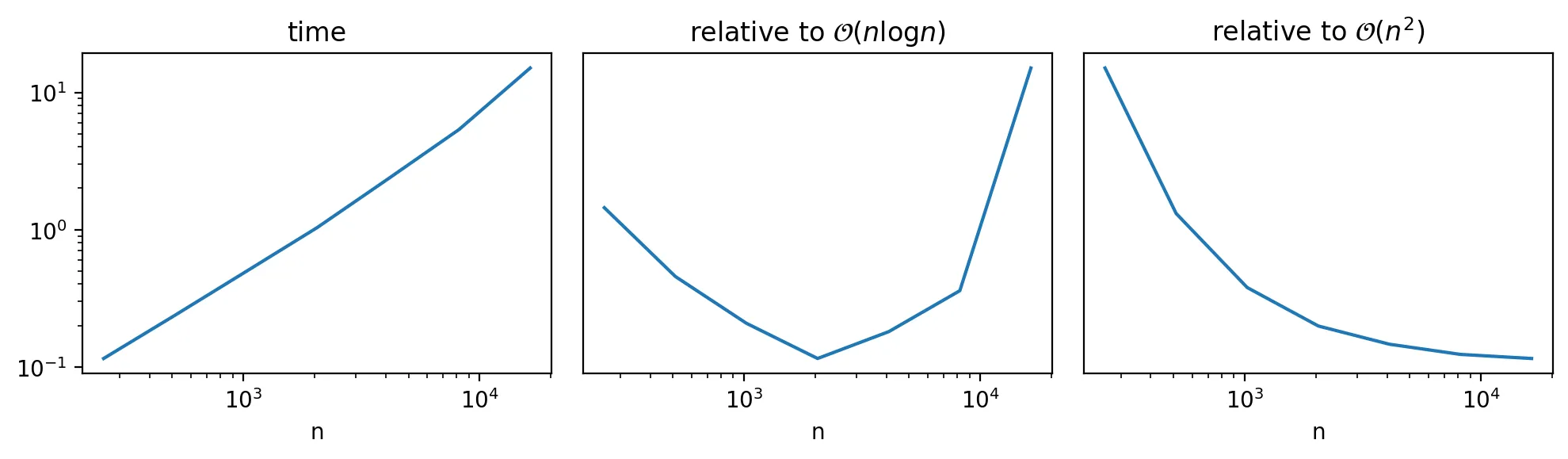
速度
使用 linkage='complete' 的层次聚类通常是 O(n^2)。但在某些情况下,可能会因为 distance_threshold 而稍微节省一些时间。为了看到这种影响,我们使用 perfplot 并探索 df 的大小与性能之间的关系:
import perfplot
tolerance = 0.005
base2_max = int(np.round(np.log2(20_000)))
o = perfplot.bench(
setup=lambda n: pd.DataFrame(np.random.uniform(0, tolerance * n, size=n), columns=['Result']),
kernels=[quantize],
n_range=[2 ** k for k in range(8, base2_max + 1)],
)
时间复杂度在 n log(n) 以上,但显然不到 n^2:
k_ = o.n_range
t_ = o.timings_s[0]
fig, axes = plt.subplots(ncols=3, figsize=(10, 3), tight_layout=True)
axes = iter(axes)
ax = next(axes)
ax.loglog(k_, t_)
ax.set_title('time')
ax.set_xlabel('n')
ax = next(axes)
ax.semilogx(k_, t_ / (np.log(k_) * k_))
ax.set_title('relative to $\mathcal{O}(n\log{}n)$')
ax.set_xlabel('n')
ax.axes.get_yaxis().set_visible(False)
ax = next(axes)
ax.semilogx(k_, t_ / k_ ** 2)
ax.set_title('relative to $\mathcal{O}(n^2)$')
ax.set_xlabel('n')
ax.axes.get_yaxis().set_visible(False)


[0, 0.006, 0.004]将产生两个组([0, 0.005]),而[0, 0.004, 0.006]将产生一个单一的组([0.0033..])。除此之外,我想到的一件事是向量量化(在1维中)。 - Pierre D