我一直在尝试使用“层次聚类”并且在R中非常简单,只需使用“hclust(as.dist(X),method="average")”。我在Python中找到了一个相当简单的方法,但是对于我的输入距离矩阵发生了一些混淆。
我有一个相似度矩阵(DF_c93tom和较小的测试版本DF_sim),我将其转换为不相似性矩阵DF_dissm = 1 - DF_sim。
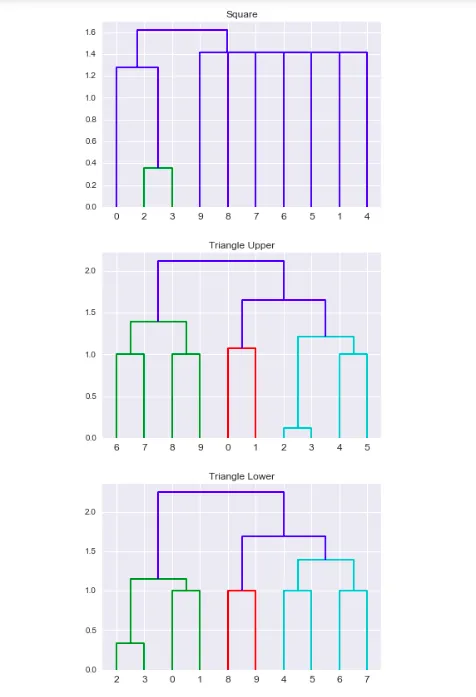
我将其用作scipy中linkage的输入,但文档说它接受一个正方形或三角形矩阵。我通过输入下三角、上三角和正方形矩阵获得了不同的聚类结果。为什么会这样?文档要求一个上三角,但下三角聚类看起来非常相似。
我的问题是,为什么所有聚类都不同?哪一个是正确的?
这是linkage的输入距离矩阵文档。
y : ndarray
A condensed or redundant distance matrix. A condensed distance matrix is a flat array containing the upper triangular of the distance matrix.
这是我的代码:
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
import pandas as pd
from scipy.cluster.hierarchy import dendrogram, linkage
%matplotlib inline
#Test Data
DF_sim = DF_c93tom.iloc[:10,:10] #Similarity Matrix
DF_sim.columns = DF_sim.index = range(10)
#print(DF_test)
# 0 1 2 3 4 5 6 7 8 9
# 0 1.000000 0 0.395833 0.083333 0 0 0 0 0 0
# 1 0.000000 1 0.000000 0.000000 0 0 0 0 0 0
# 2 0.395833 0 1.000000 0.883792 0 0 0 0 0 0
# 3 0.083333 0 0.883792 1.000000 0 0 0 0 0 0
# 4 0.000000 0 0.000000 0.000000 1 0 0 0 0 0
# 5 0.000000 0 0.000000 0.000000 0 1 0 0 0 0
# 6 0.000000 0 0.000000 0.000000 0 0 1 0 0 0
# 7 0.000000 0 0.000000 0.000000 0 0 0 1 0 0
# 8 0.000000 0 0.000000 0.000000 0 0 0 0 1 0
# 9 0.000000 0 0.000000 0.000000 0 0 0 0 0 1
#Dissimilarity Matrix
DF_dissm = 1 - DF_sim
#Redundant Matrix
#np.tril(DF_dissm).T == np.triu(DF_dissm)
#True for all values
#Hierarchical Clustering for square and triangle matrices
fig_1 = plt.figure(1)
plt.title("Square")
Z_square = linkage((DF_dissm.values),method="average")
dendrogram(Z_square)
fig_2 = plt.figure(2)
plt.title("Triangle Upper")
Z_triu = linkage(np.triu(DF_dissm.values),method="average")
dendrogram(Z_triu)
fig_3 = plt.figure(3)
plt.title("Triangle Lower")
Z_tril = linkage(np.tril(DF_dissm.values),method="average")
dendrogram(Z_tril)
plt.show()