我看过使用pymc3进行变点分析的示例,但似乎我遗漏了什么,因为我得到的结果与真实值相差甚远。这是一个玩具示例。
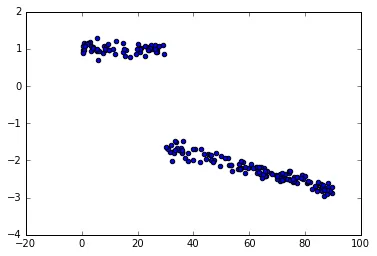
数据:
脚本:
from pymc3 import *
from numpy.random import uniform, normal
bp_u = 30 #switch point
c_u = [1, -1] #intercepts before and after switch point
beta_u = [0, -0.02] #slopes before & after switch point
x = uniform(0,90, 200)
y = (x < bp_u)*(c_u[0]+beta_u[0]*x) + (x >= bp_u)*(c_u[1]+beta_u[1]*x) + normal(0,0.1,200)
with Model() as sw_model:
sigma = HalfCauchy('sigma', beta=10, testval=1.)
switchpoint = Uniform('switchpoint', lower=x.min(), upper=x.max(), testval=45)
# Priors for pre- and post-switch intercepts and slopes
intercept_u1 = Uniform('Intercept_u1', lower=-10, upper=10)
intercept_u2 = Uniform('Intercept_u2', lower=-10, upper=10)
x_coeff_u1 = Normal('x_u1', 0, sd=20)
x_coeff_u2 = Normal('x_u2', 0, sd=20)
intercept = switch(switchpoint < x, intercept_u1, intercept_u2)
x_coeff = switch(switchpoint < x, x_coeff_u1, x_coeff_u2)
likelihood = Normal('y', mu=intercept + x_coeff * x, sd=sigma, observed=y)
start = find_MAP()
with sw_model:
step1 = NUTS([intercept_u1, intercept_u2, x_coeff_u1, x_coeff_u2])
step2 = NUTS([switchpoint])
trace = sample(2000, step=[step1, step2], start=start, progressbar=True)
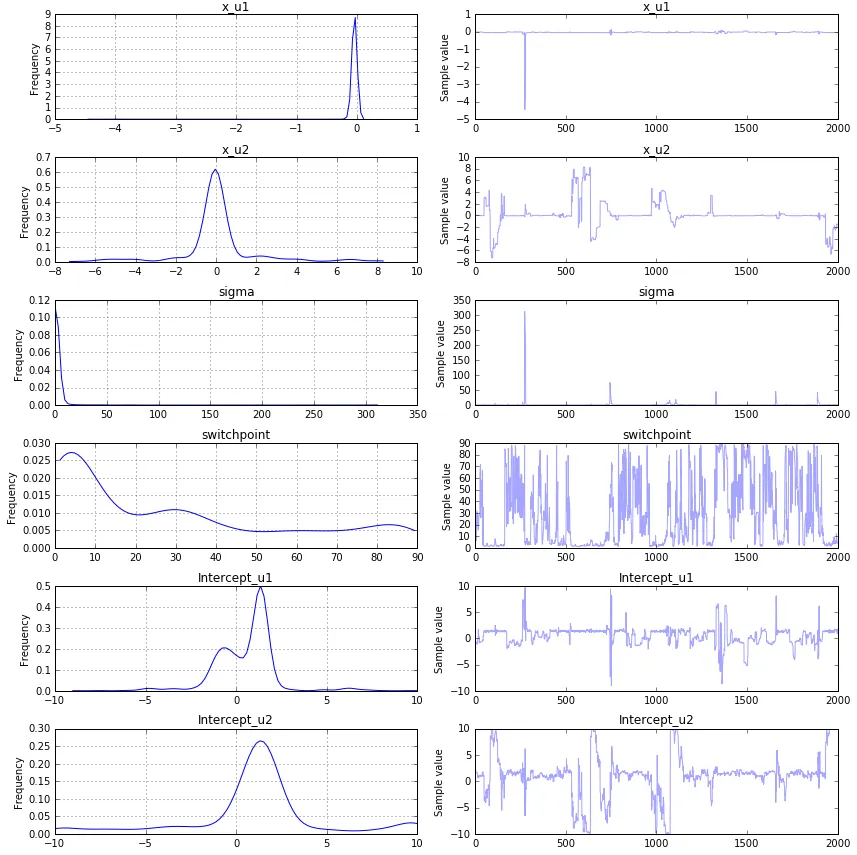
这里是结果:
正如您所看到的,它们与初始值非常不同。我做错了什么?
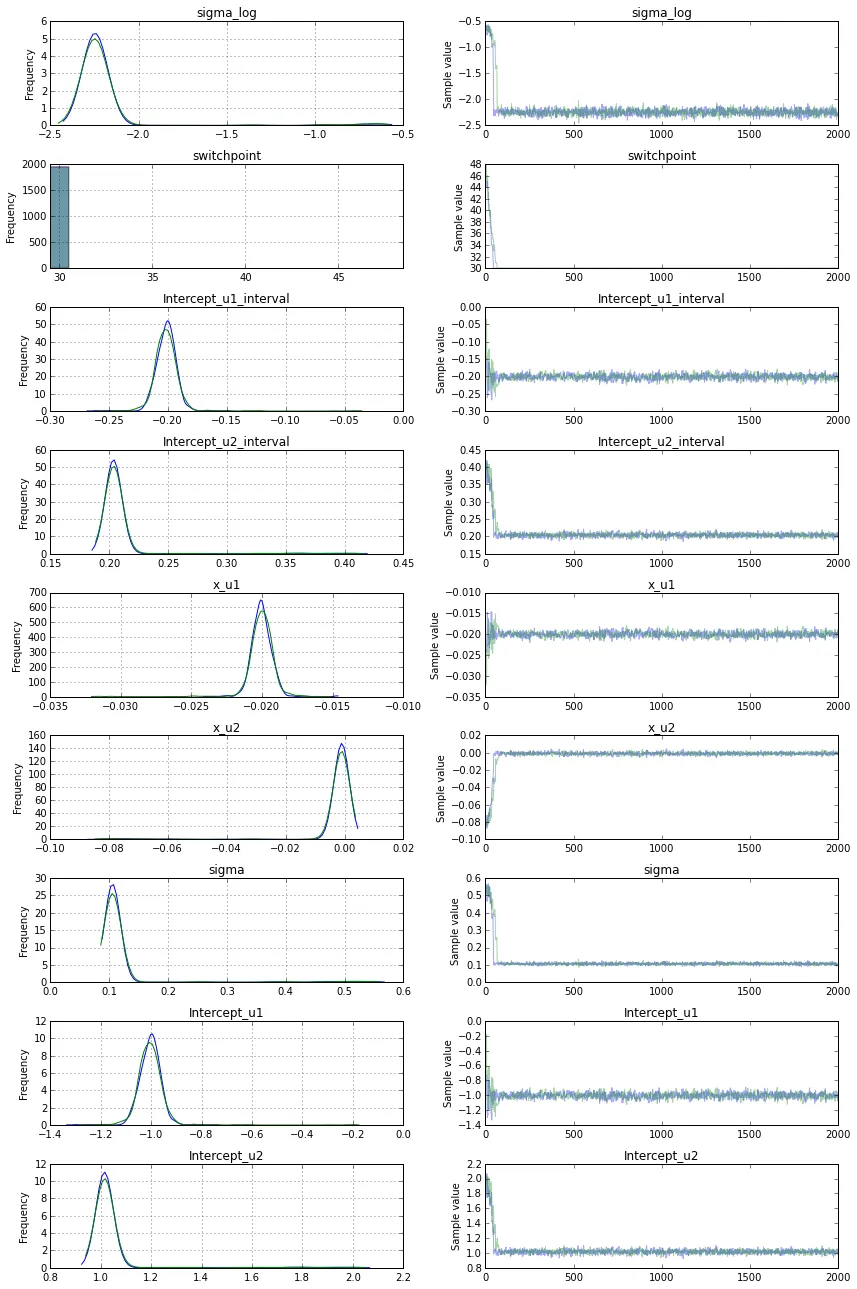
mcp包进行建模,代码为fit = mcp(list(y ~ 1, ~ 1 + x), data)。使用plot_pars(fit)函数绘制后验分布和追踪图。 - Jonas Lindeløv