我一直在尝试使用
PyMC3和
sklearn.datasets数据集中的真实数据(即不是线性函数+高斯噪声)来实现
Bayesian Linear Regression模型。我选择了属性最少的回归数据集(即
load_diabetes()),其形状为
(442,10),即
442个样本和
10个属性。我相信我的模型已经能够工作了,后验分布看起来足够好,可以尝试进行预测,以弄清楚这些东西的工作原理,但是......我意识到我不知道如何使用这些贝叶斯模型进行预测!我试图避免使用
glm和
patsy符号,因为我很难理解在使用它时实际上正在发生什么。我尝试过参考
生成pymc3推断参数的预测和
http://pymc-devs.github.io/pymc3/posterior_predictive/,但我的模型要么极其糟糕地进行预测,要么我做错了。如果我实际上正在正确进行预测(我很可能没有),那么有谁能帮助我
优化我的模型?我不知道在贝叶斯框架中是否使用最小均方误差、绝对误差或类似的东西。理想情况下,我想获得一个数组,行数=我的
X_te属性/数据测试集中的行数,列数=来自后验分布的样本数量。
import pymc3 as pm
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns; sns.set()
from scipy import stats, optimize
from sklearn.datasets import load_diabetes
from sklearn.cross_validation import train_test_split
from theano import shared
np.random.seed(9)
%matplotlib inline
diabetes_data = load_diabetes()
X, y_ = diabetes_data.data, diabetes_data.target
X_tr, X_te, y_tr, y_te = train_test_split(X,y_,test_size=0.25, random_state=0)
X.shape, y_.shape, X_tr.shape, X_te.shape
shA_X = shared(X_tr)
linear_model = pm.Model()
with linear_model:
alpha = pm.Normal("alpha", mu=0,sd=10)
betas = pm.Normal("betas", mu=0,
sd=10,
shape=X.shape[1])
sigma = pm.HalfNormal("sigma", sd=1)
mu = alpha + np.array([betas[j]*shA_X[:,j] for j in range(X.shape[1])]).sum()
likelihood = pm.Normal("likelihood", mu=mu, sd=sigma, observed=y_tr)
map_estimate = pm.find_MAP(model=linear_model, fmin=optimize.fmin_powell)
step = pm.NUTS(scaling=map_estimate)
trace = pm.sample(1000, step, start=map_estimate, progressbar=True, njobs=1)
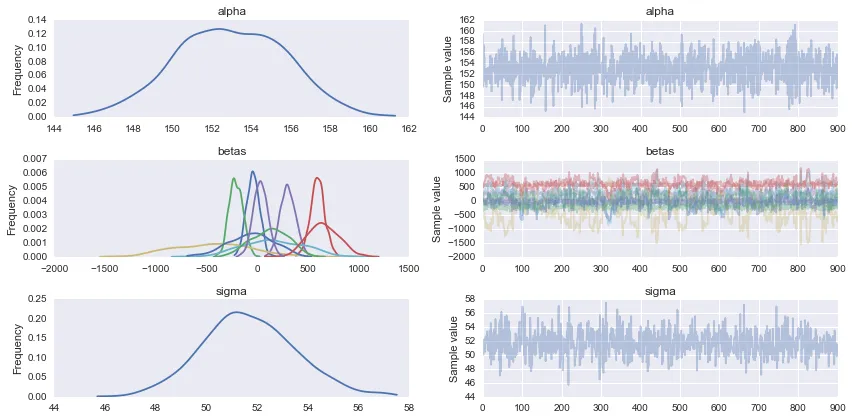
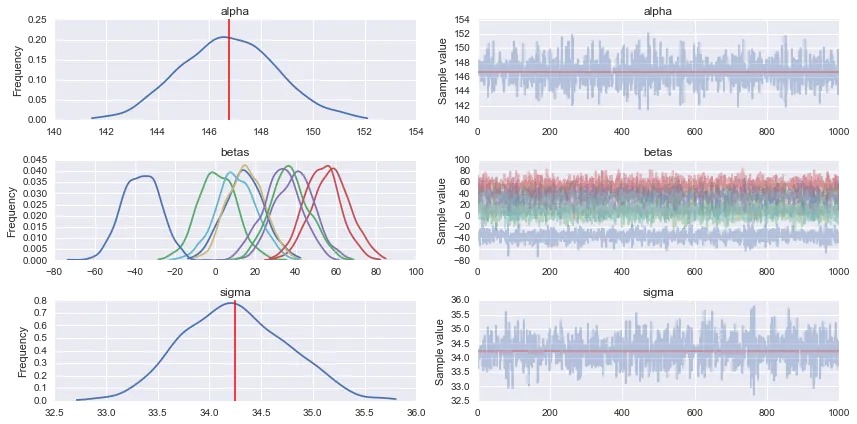
pm.traceplot(trace)
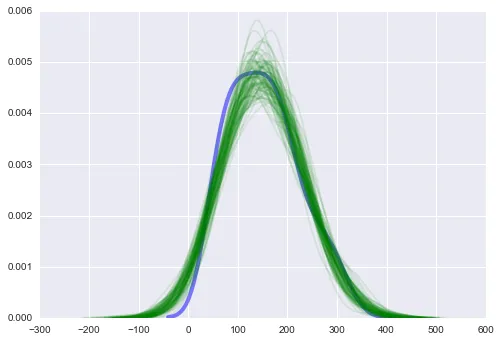
shA_X.set_value(X_te)
ppc = pm.sample_ppc(trace, model=linear_model, samples=1000)
list(ppc.items())[0][1].shape
for idx in [0,1,2,3,4,5]:
predicted_yi = list(ppc.items())[0][1].T[idx].mean()
actual_yi = y_te[idx]
print(predicted_yi, actual_yi)