我知道在R中,我可以使用tidyr来进行以下操作:
data_wide <- spread(data_protein, Fraction, Count)
data_wide将继承data_protein中未分列的所有列。
Protein Peptide Start Fraction Count
1 A 122 F1 1
1 A 122 F2 2
1 B 230 F1 3
1 B 230 F2 4
变成
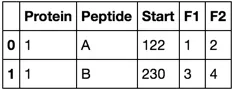
Protein Peptide Start F1 F2
1 A 122 1 2
1 B 230 3 4
但是在pandas(Python)中,
data_wide = data_prot2.reset_index(drop=True).pivot('Peptide','Fraction','Count').fillna(0)
在函数中没有指定的属性(索引,键,值)不会被继承。因此,我决定通过df.join()进行连接:
data_wide2 = data_wide.join(data_prot2.set_index('Peptide')['Start']).sort_values('Start')
但是由于存在多个起始值,这会产生肽段的重复。有没有更简单的方法来解决这个问题?或者join函数中是否有一些特殊参数可以排除重复?提前感谢您的帮助。