我希望能够查看到达rpart决策树某个节点的所有观测数据。例如,在以下代码中:
fit <- rpart(Kyphosis ~ Age + Start, data = kyphosis)
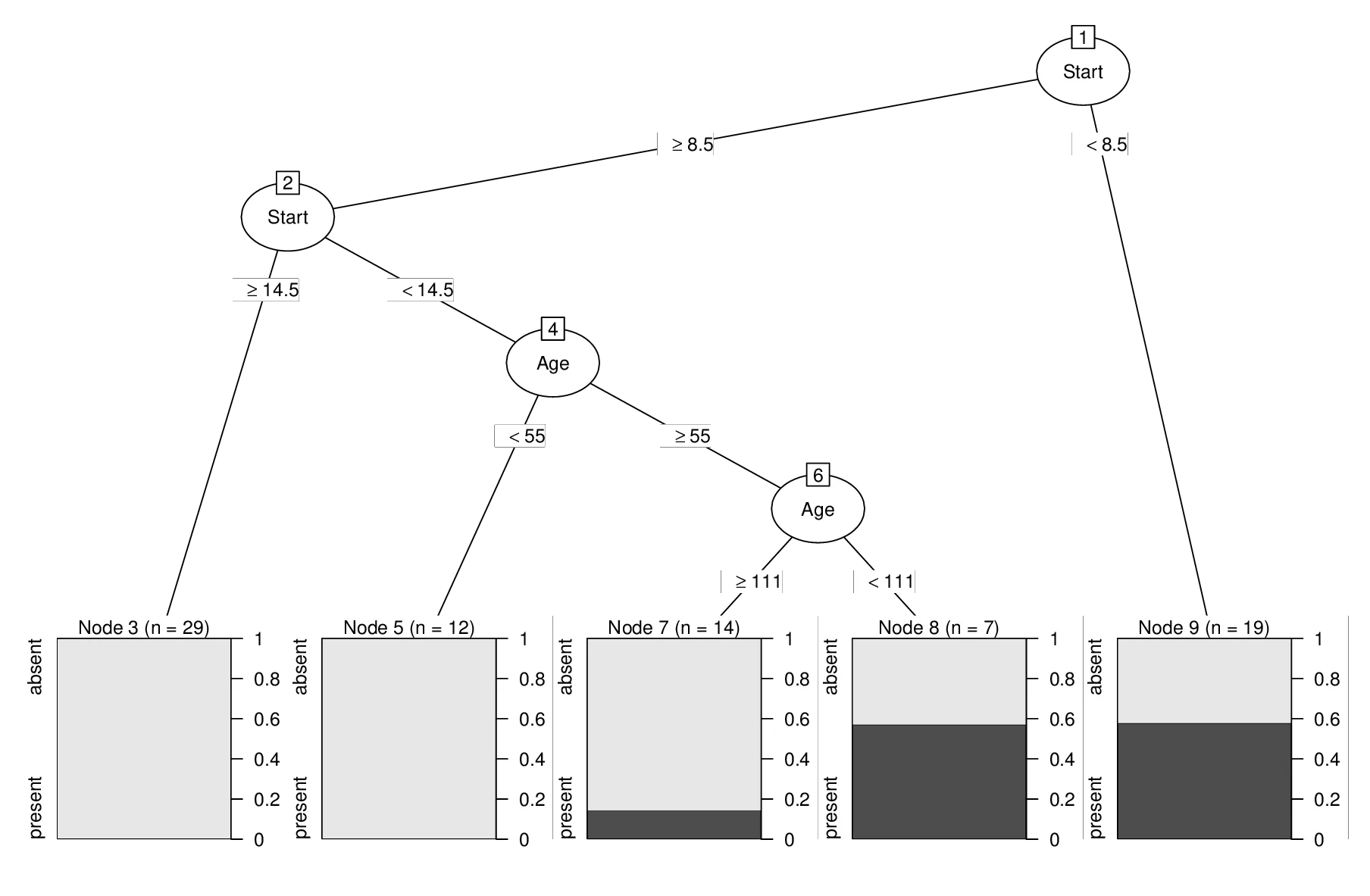
fit
n= 81
node), split, n, loss, yval, (yprob)
* denotes terminal node
1) root 81 17 absent (0.79012346 0.20987654)
2) Start>=8.5 62 6 absent (0.90322581 0.09677419)
4) Start>=14.5 29 0 absent (1.00000000 0.00000000) *
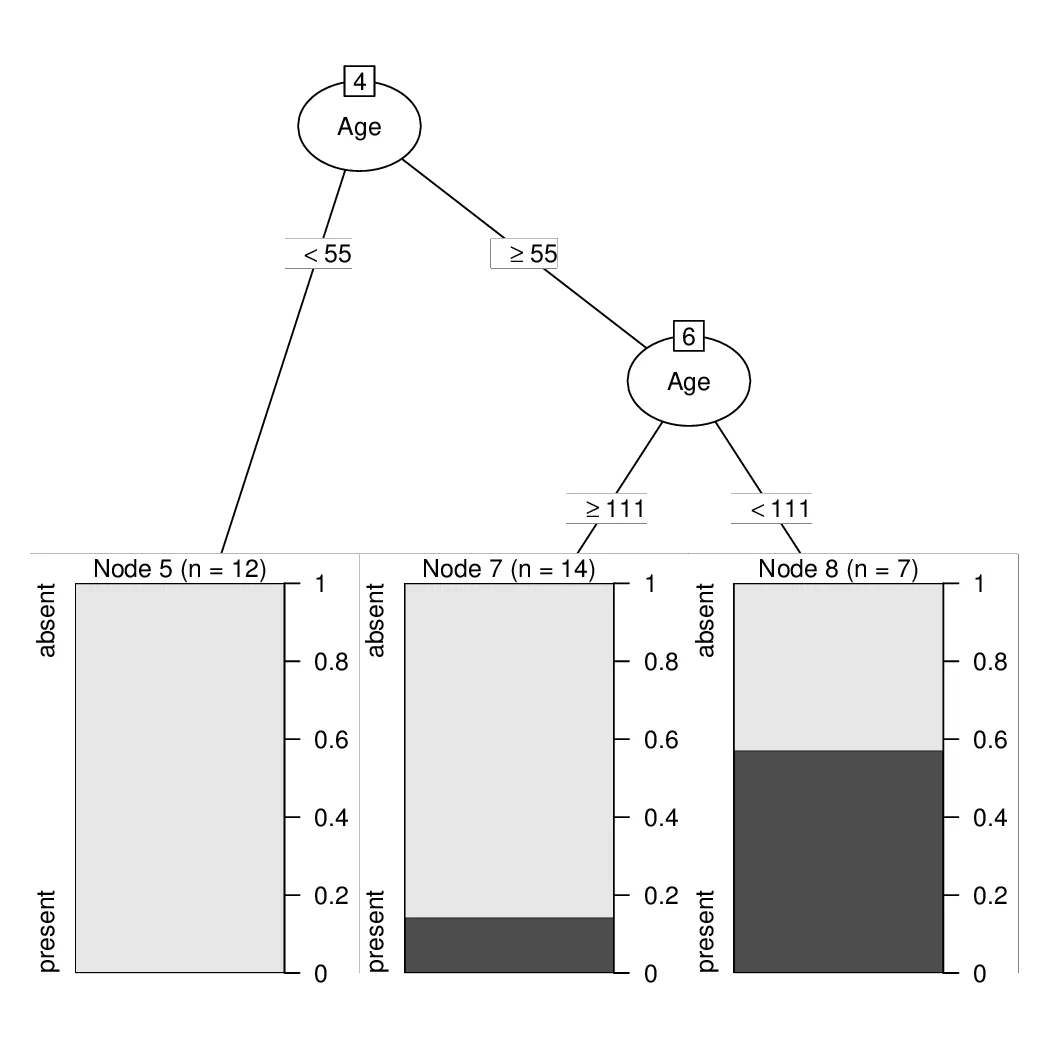
5) Start< 14.5 33 6 absent (0.81818182 0.18181818)
10) Age< 55 12 0 absent (1.00000000 0.00000000) *
11) Age>=55 21 6 absent (0.71428571 0.28571429)
22) Age>=111 14 2 absent (0.85714286 0.14285714) *
23) Age< 111 7 3 present (0.42857143 0.57142857) *
3) Start< 8.5 19 8 present (0.42105263 0.57894737) *
我希望能够查看节点(5)中的所有观测值(即:开始时间大于等于8.5且小于14.5的33个观测值)。显然,我可以手动获取它们。但我希望有一个类似于“get_node_date”的函数,只需运行get_node_date(5)即可获取相关的观测值。
你有什么建议吗?