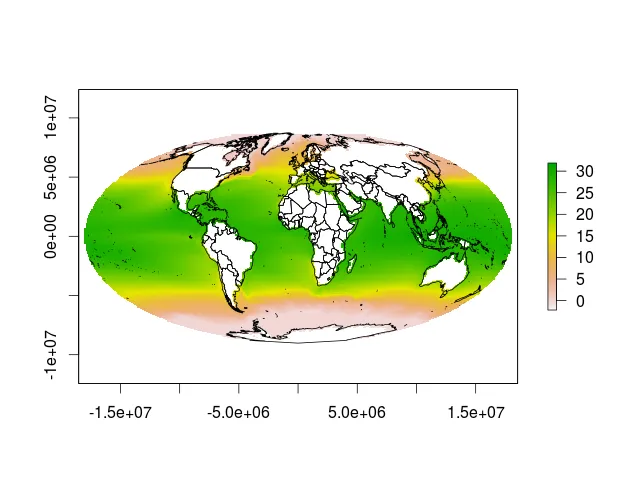
我有一个等面积Behrmann投影的光栅图像,想将其投影到Mollweide投影并绘制。
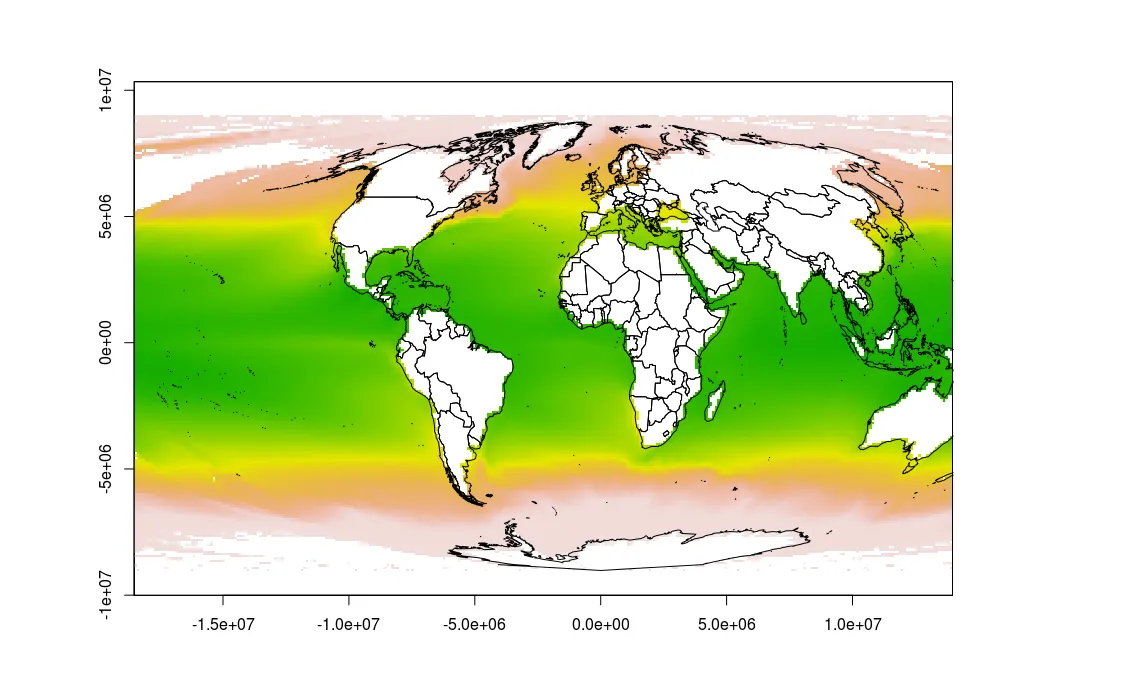
然而,当我使用以下代码进行操作时,绘图似乎不正确,因为地图延伸到了两侧,并且有各种陆地的轮廓,这些轮廓是我不希望出现的。此外,地图超出了绘图窗口。
有谁能帮我把它漂亮地绘制出来吗?
谢谢!
可以从此链接下载使用的数据文件。
这是我目前拥有的代码:
require(rgdal)
require(maptools)
require(raster)
data(wrld_simpl)
mollCRS <- CRS('+proj=moll')
behrmannCRS <- CRS('+proj=cea +lat_ts=30')
sst <- raster("~/Dropbox/Public/sst.tif", crs=behrmannCRS)
sst_moll <- projectRaster(sst, crs=mollCRS)
wrld <- spTransform(wrld_simpl, mollCRS)
plot(sst_moll)
plot(wrld, add=TRUE)