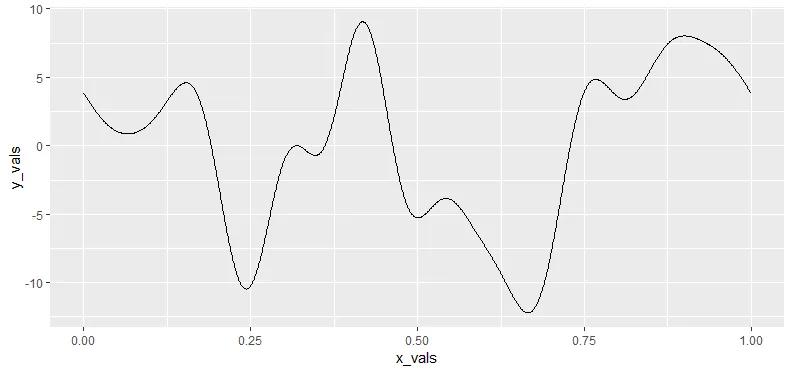
应用傅里叶变换到脑电图测量数据后,我想将FFT的近似值与原始信号以图形形式进行比较。我需要将FFT的数据(频率和强度)转换回时间序列。
为了转换原始时间序列,我使用eegfft方法中的eegkit包。我得到一系列频率和振幅来近似原始信号。
这里展示了FFT的两个结果作为缩短的示例:
有没有一种包或方法可以从通过FFT近似的频率和振幅数据中重建时间序列?
这里展示了FFT的两个结果作为缩短的示例:
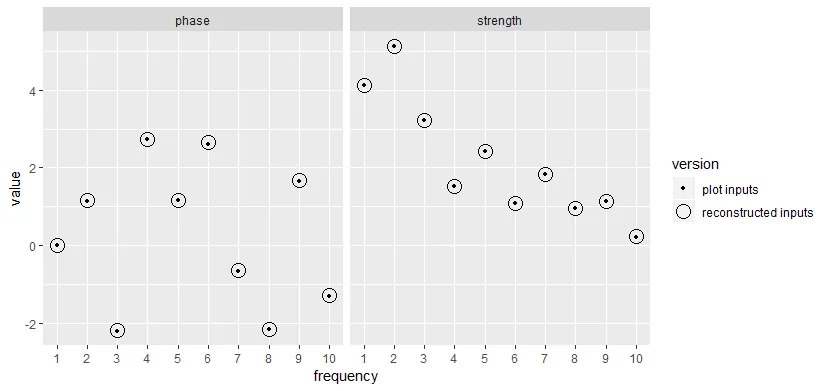
# Frequency in Hz
freq <- c(1, 2, 3, 4, 5, 6, 7, 8, 9, 10)
# Strength in uV
ampl <- c(4.1135352, 5.1272713, 3.2069741, 1.5336438, 2.4301334, 1.0974758, 1.8238327, 0.9637886, 1.1401306, 0.2224472)
有没有一种包或方法可以从通过FFT近似的频率和振幅数据中重建时间序列?
编辑:
对于原始信号的重构,我是否还需要eegfft方法返回的相位信息?
# Phase shift in range -pi to pi
phase <- c(0.0000000, 1.1469542, -2.1930702, 2.7361738,1.1597980, 2.6118647, -0.6609641, -2.1508755,1.6584852, -1.2906986)