我正在尝试使用Python(最好是使用matplotlib和networkx,尽管我对bokeh也很感兴趣)创建类似于下面的线性网络图。
如何使用Python中的networkx高效地构建此图形绘制(pos)? 我想在更复杂的示例中使用它,因此我觉得为这个简单的示例硬编码位置将没有用处:(。是否有适用于此的networkx解决方案? pos(字典,可选)-一个以节点为键、位置为值的字典。如果未指定,则会计算弹簧布局定位。有关计算节点位置的函数,请参见networkx.layout。
我还没有看到任何关于如何在
我还没有看到任何关于如何在
networkx中实现此目标的教程,这就是为什么我相信这个问题将成为社区可靠的资源。我已经广泛地阅读了networkx教程,但是没有类似的内容。 networkx的布局会使这种类型的网络难以解释,除非仔细使用pos参数...这是我唯一的选择。 https://networkx.github.io/documentation/networkx-1.9/reference/drawing.html文档中预先计算的布局似乎都无法很好地处理这种类型的网络结构。
简单例子:
(A)每个外部键表示从左到右移动的图表中的迭代(例如,迭代0表示样本,迭代1具有组1-3,迭代2相同,迭代3具有组1-2等)。 (B)内部字典包含该特定迭代中的当前分组以及表示当前组的先前组合的权重(例如,迭代3具有Group 1和Group 2,对于迭代4,所有迭代3的Group 2都进入了迭代4的Group 2,但迭代3的Group 1已被拆分。权重总和始终为1。
我用于上面图形连接带权重的代码:
D_iter_current_previous = {
1: {
"Group 1":{"sample_0":0.5, "sample_1":0.5, "sample_2":0, "sample_3":0, "sample_4":0},
"Group 2":{"sample_0":0, "sample_1":0, "sample_2":1, "sample_3":0, "sample_4":0},
"Group 3":{"sample_0":0, "sample_1":0, "sample_2":0, "sample_3":0.5, "sample_4":0.5}
},
2: {
"Group 1":{"Group 1":1, "Group 2":0, "Group 3":0},
"Group 2":{"Group 1":0, "Group 2":1, "Group 3":0},
"Group 3":{"Group 1":0, "Group 2":0, "Group 3":1}
},
3: {
"Group 1":{"Group 1":0.25, "Group 2":0, "Group 3":0.75},
"Group 2":{"Group 1":0.25, "Group 2":0.75, "Group 3":0}
},
4: {
"Group 1":{"Group 1":1, "Group 2":0},
"Group 2":{"Group 1":0.25, "Group 2":0.75}
}
}
当我使用 networkx 制作图形时发生了以下情况:
import networkx
import matplotlib.pyplot as plt
# Create Directed Graph
G = nx.DiGraph()
# Iterate through all connections
for iter_n, D_current_previous in D_iter_current_previous.items():
for current_group, D_previous_weights in D_current_previous.items():
for previous_group, weight in D_previous_weights.items():
if weight > 0:
# Define connections using `|__|` as a delimiter for the names
previous_node = "%d|__|%s"%(iter_n - 1, previous_group)
current_node = "%d|__|%s"%(iter_n, current_group)
connection = (previous_node, current_node)
G.add_edge(*connection, weight=weight)
# Draw Graph with labels and width thickness
nx.draw(G, with_labels=True, width=[G[u][v]['weight'] for u,v in G.edges()])
matplotlib中创建一个散点图,每个刻度代表一个迭代(包括初始样本),然后用不同的权重将点连接起来。这将是一些相当混乱的代码,尤其是尝试将标记的边缘与连接线对齐...但是,我不确定这和networkx是最好的方法,还是有一个专门用于这种绘图的工具(例如bokeh或plotly)。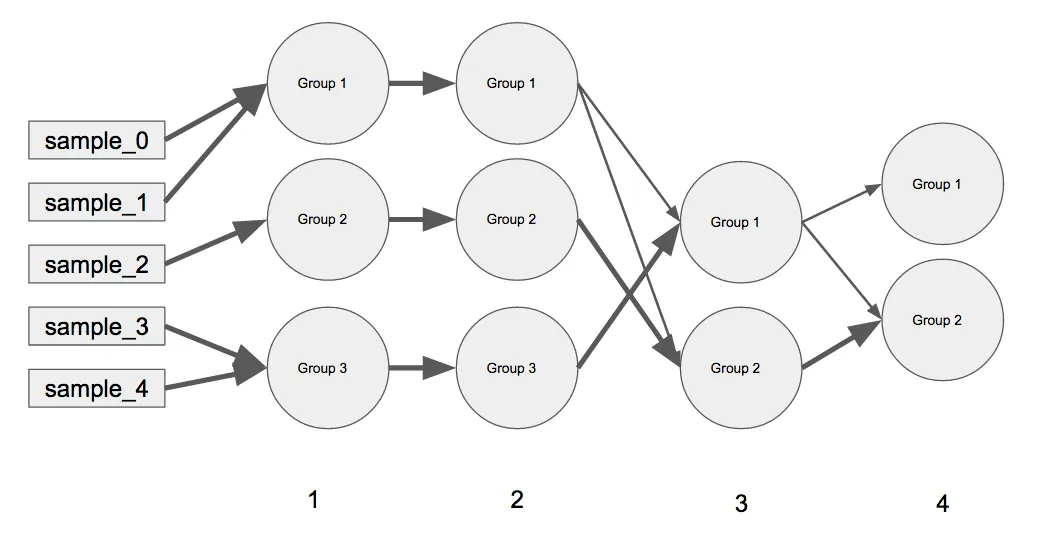
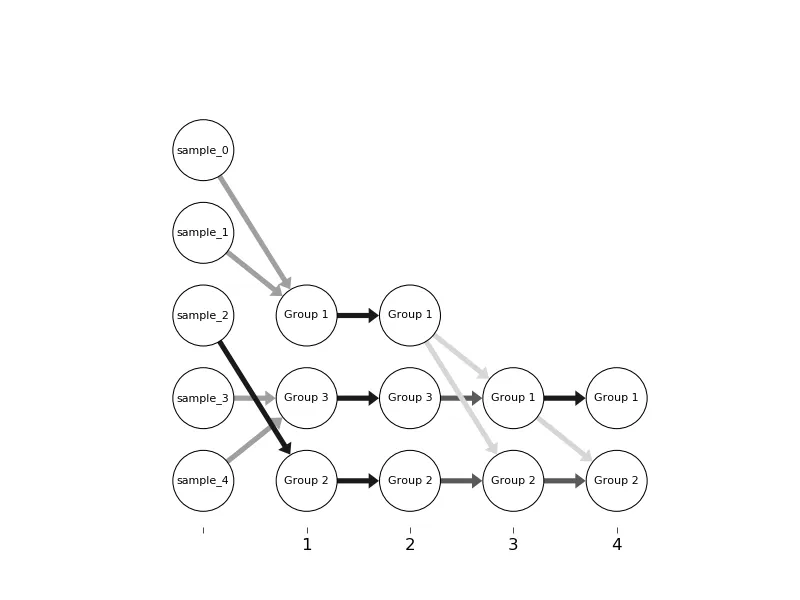
prog ='dot'至关重要,但可能会帮助你达到目标。请注意,除了networkx和matplotlib之外,还需要安装graphviz和pygraphviz。 - J Richard Snape