假设我的正类变量是“购买”,负类变量是“不购买”,我有一个名为“dt.train”的训练集和一个单独的测试集“dt.test”。
在普通决策树中,对于新数据行(测试数据),“购买”类别的类别概率P(has_bought="buy")是通过将所有具有“购买”类别的叶子节点中的观测数据数除以该叶子节点的总观测数据数(基于用于生成该树的训练数据)来计算的。
然而,h2o.gbm似乎做了一些不同的事情,即使我模拟了“正常”的决策树(将n.trees设置为1,并将所有sample.rate参数设置为1)。我认为最好的方法是逐步说明这种混淆。 第1步:训练模型 我不关心过度拟合或模型性能。我想让自己的工作尽可能简单,所以将n.trees设置为1,并确保每个树和分裂都使用了所有的训练数据(行和列),通过将所有的sample.rate参数设置为1。以下是训练模型的代码。
base.gbm.model <- h2o.gbm(
x = predictors,
y = "has_bought",
training_frame = dt.train,
model_id = "2",
nfolds = 0,
ntrees = 1,
learn_rate = 0.001,
max_depth = 15,
sample_rate = 1,
col_sample_rate = 1,
col_sample_rate_per_tree = 1,
seed = 123456,
keep_cross_validation_predictions = TRUE,
stopping_rounds = 10,
stopping_tolerance = 0,
stopping_metric = "AUC",
score_tree_interval = 0
)
第二步:获取训练集的叶子节点分配
我的目的是使用用于训练模型的相同数据,并了解它们最终停留在哪个叶子节点。H2o提供了一个函数来完成这个任务,如下所示。
train.leafs <- h2o.predict_leaf_node_assignment(base.gbm.model, dt.train)
这将返回训练数据每行的叶结点分配(例如“LLRRLL”)。由于我们只有一棵树,因此该列称为“T1.C1”,我将其重命名为“leaf_node”,然后与训练数据的目标变量“has_bought”进行绑定。这将导致以下输出(从现在开始称为“train.leafs”)。
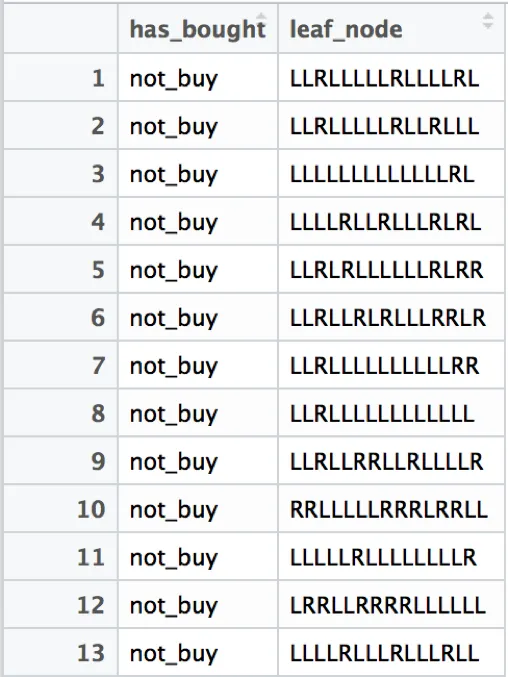
步骤3:对测试集进行预测
对于测试集,我想要预测两个结果:
- The prediction of the model itself P(has_bought="buy")
The leaf node assignment according to the model.
test.leafs <- h2o.predict_leaf_node_assignment(base.gbm.model, dt.test) test.pred <- h2o.predict(base.gbm.model, dt.test)
test.total <- h2o.cbind(dt.test[, c("has_bought")], test.pred, test.leafs)
其结果如下表所示,下文将其称为“test.total”。
不幸的是,我没有足够的声望来发布超过2个链接。但如果您在第5步中点击“与手动概率计算相结合的“test.total”表”,它基本上是相同的表格,只是没有“manual_prob_buy”列。
第4步:手动预测概率
理论上,现在我应该能够自己预测概率了。我编写了一个循环,循环遍历“test.total”中的每一行。对于每一行,我采用叶节点分配。
然后,我使用该叶节点分配来过滤“train.leafs”表,并检查在与测试行相关联的叶子中有多少观察值具有正类(has_bought == 1)(posN),以及总共有多少观察值(totalN)。
我执行(标准)计算 posN / totalN,并将其存储在测试行中作为名为“manual_prob_buy”的新列,这应该是该叶子的P(has_bought="buy")的概率。因此,落在此叶子中的每个测试行都应获得此概率。以下是此for循环的示例。
for(i in 1:nrow(dt.test)){
leaf <- test.total[i, leaf_node]
totalN <- nrow(train.leafs[train.leafs$leaf_node == leaf])
posN <- nrow(train.leafs[train.leafs$leaf_node == leaf & train.leafs$has_bought == "buy",])
test.total[i, manual_prob_buy := posN / totalN]
}
第5步:比较概率
这是我感到困惑的地方。以下是更新后的“test.total”表,其中“buy”表示模型中的P(has_bought =“buy”)概率,“manual_prob_buy”表示从第4步手动计算得出的概率。据我所知,这些概率应该相同,因为我只使用了1棵树,并且将sample.rates设置为1。
“test.total”表与手动概率计算相结合
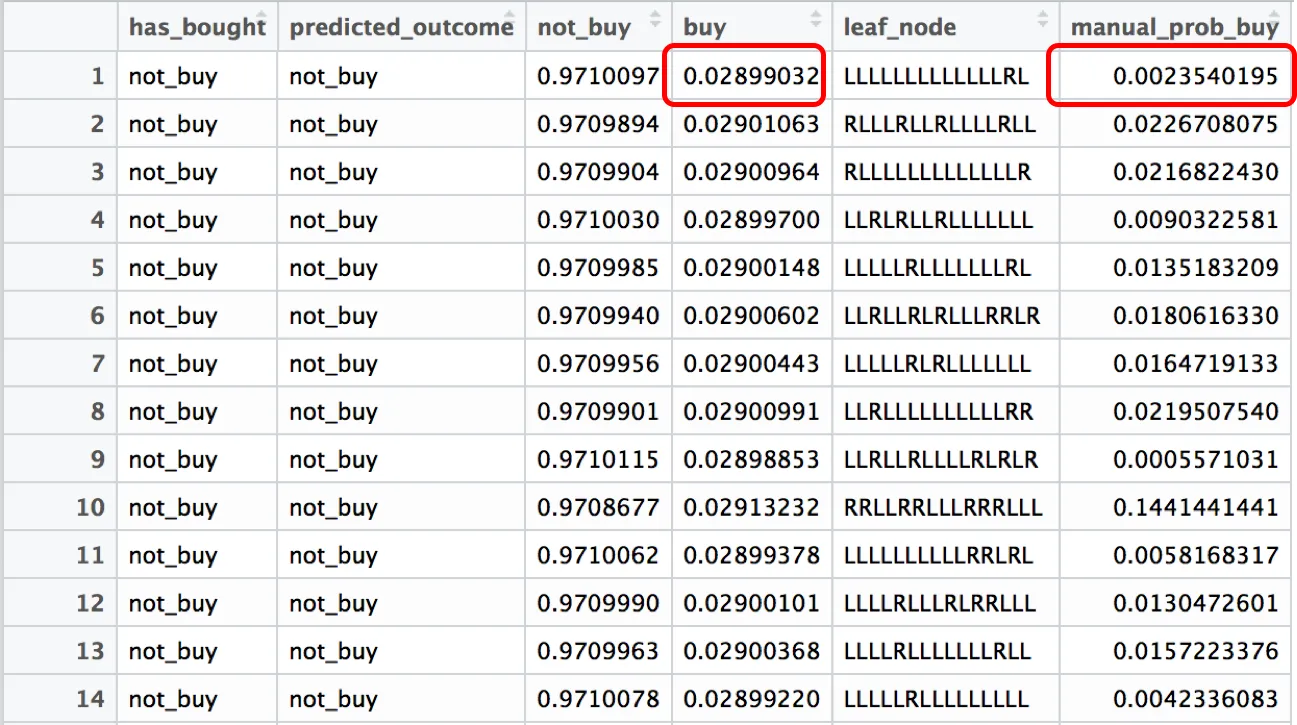 问题:
问题:我不理解为什么这两个概率不一样。据我所知,我已经以普通分类树的方式设置了参数。
因此问题就是:有没有人知道为什么我在这些概率中发现差异?
我希望有人能指出我可能犯了哪些错误,我真的希望自己只是做了一些愚蠢的事情,因为这让我抓狂了。
谢谢!