这个数学函数的目的是使用二面角计算两个(或多个)蛋白质结构之间的距离:
它在结构生物学中非常有用。我已经用numpy编写了这个函数,但目标是拥有更快的实现。作为计算时间参考,我使用了scikit-learn包中可用的欧几里德距离函数。
这是我目前的代码:
import numpy as np
import numexpr as ne
from sklearn.metrics.pairwise import euclidean_distances
# We have 10000 structures with 100 dihedral angles
n = 10000
m = 100
# Generate some random data
c = np.random.rand(n,m)
# Generate random int number
x = np.random.randint(c.shape[0])
print c.shape, x
# First version with numpy of the dihedral_distances function
def dihedral_distances(a, b):
l = 1./a.shape[0]
return np.sqrt(l* np.sum((0.5)*(1. - np.cos(a-b)), axis=1))
# Accelerated version with numexpr
def dihedral_distances_ne(a, b):
l = 1./a.shape[0]
tmp = ne.evaluate('sum((0.5)*(1. - cos(a-b)), axis=1)')
return ne.evaluate('sqrt(l* tmp)')
# The function of reference I try to be close as possible
# in term of computation time
%timeit euclidean_distances(c[x,:], c)[0]
1000 loops, best of 3: 1.07 ms per loop
# Computation time of the first version of the dihedral_distances function
# We choose randomly 1 structure among the 10000 structures.
# And we compute the dihedral distance between this one and the others
%timeit dihedral_distances(c[x,:], c)
10 loops, best of 3: 21.5 ms per loop
# Computation time of the accelerated function with numexpr
%timeit dihedral_distances_ne(c[x,:], c)
100 loops, best of 3: 9.44 ms per loop
9.44毫秒非常快,但如果需要运行一百万次,它就非常慢。现在的问题是,下一步该怎么做?是Cython?还是PyOpenCL?我有一些PyOpenCL的经验,但我从未编写过像这样复杂的代码。我不知道是否可以像使用numpy那样在GPU上一次计算二面角距离,并且如何继续操作。
谢谢你的帮助!
编辑: 谢谢大家!我正在全力解决方案,一旦完成我会把代码放在这里。
CYTHON版本:
%load_ext cython
import numpy as np
np.random.seed(1234)
n = 10000
m = 100
c = np.random.rand(n,m)
x = np.random.randint(c.shape[0])
print c.shape, x
%%cython --compile-args=-fopenmp --link-args=-fopenmp --force
import numpy as np
cimport numpy as np
from libc.math cimport sqrt, cos
cimport cython
from cython.parallel cimport parallel, prange
# Define a function pointer to a metric
ctypedef double (*metric)(double[: ,::1], np.intp_t, np.intp_t)
cdef extern from "math.h" nogil:
double cos(double x)
double sqrt(double x)
@cython.boundscheck(False)
@cython.wraparound(False)
@cython.cdivision(True)
cdef double dihedral_distances(double[:, ::1] a, np.intp_t i1, np.intp_t i2):
cdef double res
cdef int m
cdef int j
res = 0.
m = a.shape[1]
for j in range(m):
res += 1. - cos(a[i1, j] - a[i2, j])
res /= 2.*m
return sqrt(res)
@cython.boundscheck(False)
@cython.wraparound(False)
@cython.cdivision(True)
cdef double dihedral_distances_p(double[:, ::1] a, np.intp_t i1, np.intp_t i2):
cdef double res
cdef int m
cdef int j
res = 0.
m = a.shape[1]
with nogil, parallel(num_threads=2):
for j in prange(m, schedule='dynamic'):
res += 1. - cos(a[i1, j] - a[i2, j])
res /= 2.*m
return sqrt(res)
@cython.boundscheck(False)
@cython.wraparound(False)
def pairwise(double[: ,::1] c not None, np.intp_t x, p = True):
cdef metric dist_func
if p:
dist_func = &dihedral_distances_p
else:
dist_func = &dihedral_distances
cdef np.intp_t i, n_structures
n_samples = c.shape[0]
cdef double[::1] res = np.empty(n_samples)
for i in range(n_samples):
res[i] = dist_func(c, x, i)
return res
%timeit pairwise(c, x, False)
100 loops, best of 3: 17 ms per loop
# Parallel version
%timeit pairwise(c, x, True)
10 loops, best of 3: 37.1 ms per loop
所以我跟着你的链接创建了dihedral distances函数的Cython版本。我们获得了一些速度提升,虽然不是很多,但仍然比numexpr版本慢(17毫秒对9.44毫秒)。所以我尝试使用prange并行化该函数,但结果更差(37.1毫秒对17毫秒对9.4毫秒)!
我错过了什么吗?
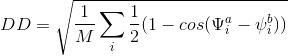
*0.5放在求和符号外面;2)在从1中减去之前对cos进行求和(这样做更准确,因为总和会接近于1)。对我来说,这些改进将运行时间从25毫秒缩短到了17毫秒。我知道你正在寻找更多的改进,但这是我的全部建议,希望能有所帮助。 - tom10sqrt(sum(1 - cos(x)))就变成了sqrt(N - sum(cos(x)))。你记得N了吗? - tom10