我已经在网上搜索过,但仍然没有找到答案或方法来解决以下问题
我正在将一些MATLAB代码翻译成Python,在MATLAB中,我希望使用以下函数找到核密度估计:
[p,x] = ksdensity(data)
其中p是分布中点x的概率。
Scipy有一个函数,但是只返回p。
有没有办法找到x值处的概率?
谢谢!
ksdensity 调用会自动生成一个任意的 x。而 scipy.stats.gaussian_kde() 则返回一个可调用函数,它可以使用您选择的任何 x 进行评估。等价的 x 是 np.linspace(data.min(), data.max(), 100)。import numpy as np
from scipy import stats
data = ...
kde = stats.gaussian_kde(data)
x = np.linspace(data.min(), data.max(), 100)
p = kde(x)
另一种选择是使用Scikit-Learn Python包中的核密度估计器,sklearn.neighbors.KernelDensity
以下是一个类似于Matlab文档ksdensity的高斯分布示例:
import numpy as np
import matplotlib.pyplot as plt
from sklearn.neighbors import KernelDensity
np.random.seed(12345)
# similar to MATLAB ksdensity example x = [randn(30,1); 5+randn(30,1)];
Vecvalues=np.concatenate((np.random.normal(0,1,30), np.random.normal(5,1,30)))[:,None]
Vecpoints=np.linspace(-8,12,100)[:,None]
kde = KernelDensity(kernel='gaussian', bandwidth=0.5).fit(Vecvalues)
logkde = kde.score_samples(Vecpoints)
plt.plot(Vecpoints,np.exp(logkde))
plt.show()
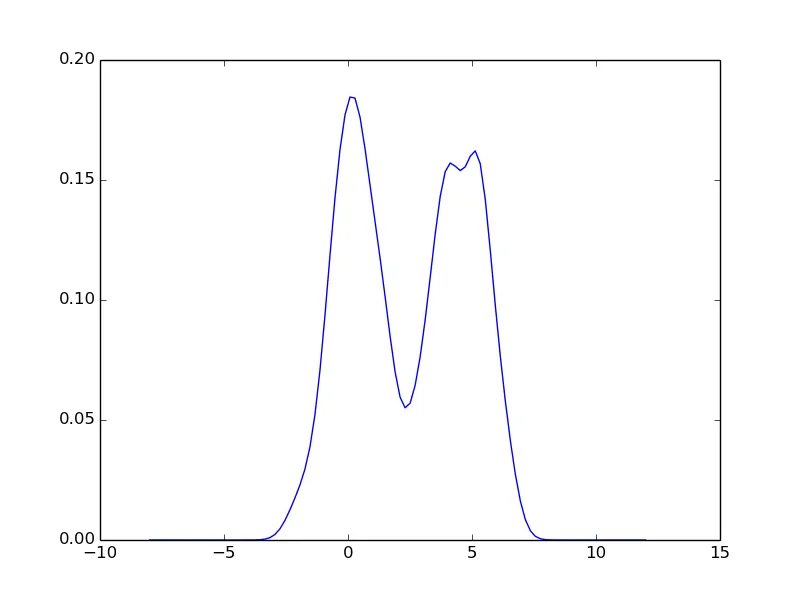
scipy.stats.gaussian_kde()和sklearn.neighbors.KernelDensity两种方法,第一种出现了“核心死亡”的错误,而第二种方法可以正常工作。 - ZK Zhao