对于以下2016年全球每日海表面温度的netcdf文件,我试图 (i) 在时间上进行子集,(ii) 在地理上进行子集,(iii) 然后对每个像素取长期平均值并创建一个基本图。
文件链接: 这里
现在我想只选取9月1日到10月15日的数据子集,但不知道如何实现...
在进行时间子集操作之后,创建栅格砖块(或堆栈)和地理子集。
最后,我希望对所有数据中的每个像素进行平均,并将其分配给单个光栅,然后制作一个简单的图表...感谢任何帮助和指导。
文件链接: 这里
library(raster)
library(ncdf4)
设置工作目录后打开netcdf文件。
nc_data <- nc_open('sst.day.mean.2016.v2.nc')
更改时间变量,使其易于解释。
time <- ncdf4::ncvar_get(nc_data, varid="time")
head(time)
将日期更改为我可以解释的格式
time_d <- as.Date(time, format="%j", origin=as.Date("1800-01-01"))
现在我想只选取9月1日到10月15日的数据子集,但不知道如何实现...
在进行时间子集操作之后,创建栅格砖块(或堆栈)和地理子集。
b <- brick('sst.day.mean.2016.v2.nc') # I would change this name to my file with time subest
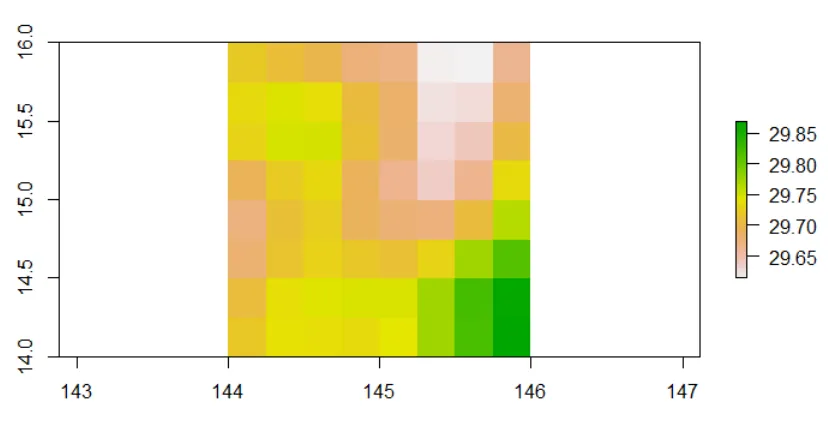
地理子集
b <- crop(b, extent(144, 146, 14, 16))
最后,我希望对所有数据中的每个像素进行平均,并将其分配给单个光栅,然后制作一个简单的图表...感谢任何帮助和指导。