问题: 是否有一种方法可以从randomForest对象中提取每个单独的CART模型的变量重要性?
rf_mod$forest似乎没有这个信息,文档也没有提及。
在R的
randomForest包中,整个CART模型森林的平均变量重要性可以通过importance(rf_mod)来获取。library(randomForest)
df <- mtcars
set.seed(1)
rf_mod = randomForest(mpg ~ .,
data = df,
importance = TRUE,
ntree = 200)
importance(rf_mod)
%IncMSE IncNodePurity
cyl 6.0927875 111.65028
disp 8.7730959 261.06991
hp 7.8329831 212.74916
drat 2.9529334 79.01387
wt 7.9015687 246.32633
qsec 0.7741212 26.30662
vs 1.6908975 31.95701
am 2.5298261 13.33669
gear 1.5512788 17.77610
carb 3.2346351 35.69909
我们也可以使用
getTree来提取单个树结构。这是第一棵树。head(getTree(rf_mod, k = 1, labelVar = TRUE))
left daughter right daughter split var split point status prediction
1 2 3 wt 2.15 -3 18.91875
2 0 0 <NA> 0.00 -1 31.56667
3 4 5 wt 3.16 -3 17.61034
4 6 7 drat 3.66 -3 21.26667
5 8 9 carb 3.50 -3 15.96500
6 0 0 <NA> 0.00 -1 19.70000
一种解决方法是多生成CARTs(即ntree = 1),获取每个树的变量重要性,并平均得到结果的%IncMSE:
# number of trees to grow
nn <- 200
# function to run nn CART models
run_rf <- function(rand_seed){
set.seed(rand_seed)
one_tr = randomForest(mpg ~ .,
data = df,
importance = TRUE,
ntree = 1)
return(one_tr)
}
# list to store output of each model
l <- vector("list", length = nn)
l <- lapply(1:nn, run_rf)
提取、平均和比较步骤。
# extract importance of each CART model
library(dplyr); library(purrr)
map(l, importance) %>%
map(as.data.frame) %>%
map( ~ { .$var = rownames(.); rownames(.) <- NULL; return(.) } ) %>%
bind_rows() %>%
group_by(var) %>%
summarise(`%IncMSE` = mean(`%IncMSE`)) %>%
arrange(-`%IncMSE`)
# A tibble: 10 x 2
var `%IncMSE`
<chr> <dbl>
1 wt 8.52
2 cyl 7.75
3 disp 7.74
4 hp 5.53
5 drat 1.65
6 carb 1.52
7 vs 0.938
8 qsec 0.824
9 gear 0.495
10 am 0.355
# compare to the RF model above
importance(rf_mod)
%IncMSE IncNodePurity
cyl 6.0927875 111.65028
disp 8.7730959 261.06991
hp 7.8329831 212.74916
drat 2.9529334 79.01387
wt 7.9015687 246.32633
qsec 0.7741212 26.30662
vs 1.6908975 31.95701
am 2.5298261 13.33669
gear 1.5512788 17.77610
carb 3.2346351 35.69909
我希望能够直接从
randomForest对象中提取每个树的变量重要性,而不需要通过完全重新运行RF来实现可再现的累积变量重要性图,如此一类,以及下面显示的mtcars的图表。这里是最小示例。我知道单棵树的变量重要性并没有统计学意义,并且我也不想孤立地解释树。我需要它们来进行可视化和传达的信息是:随着森林中的树增加,变量重要性度量会在稳定之前跳动。
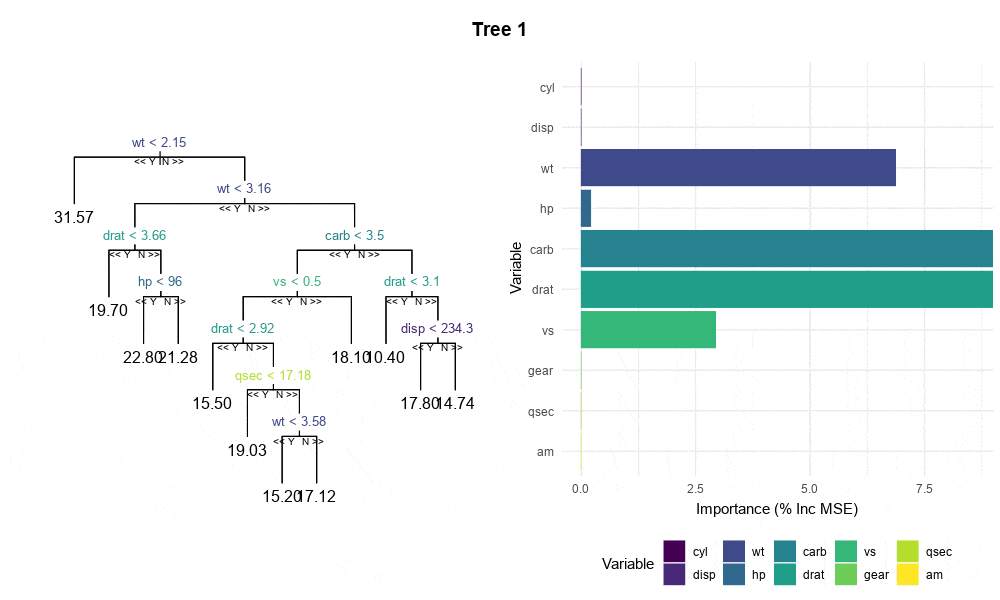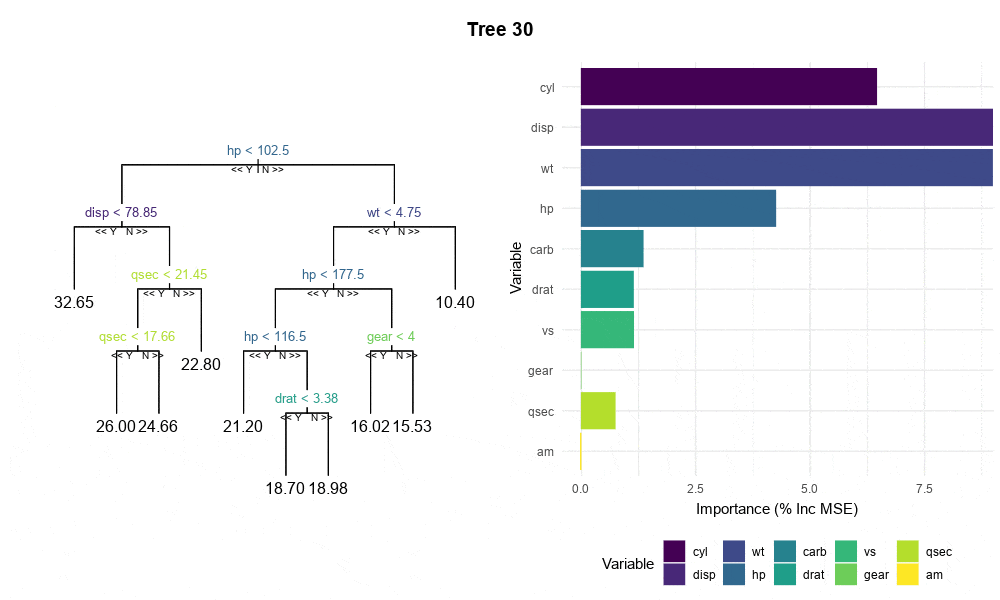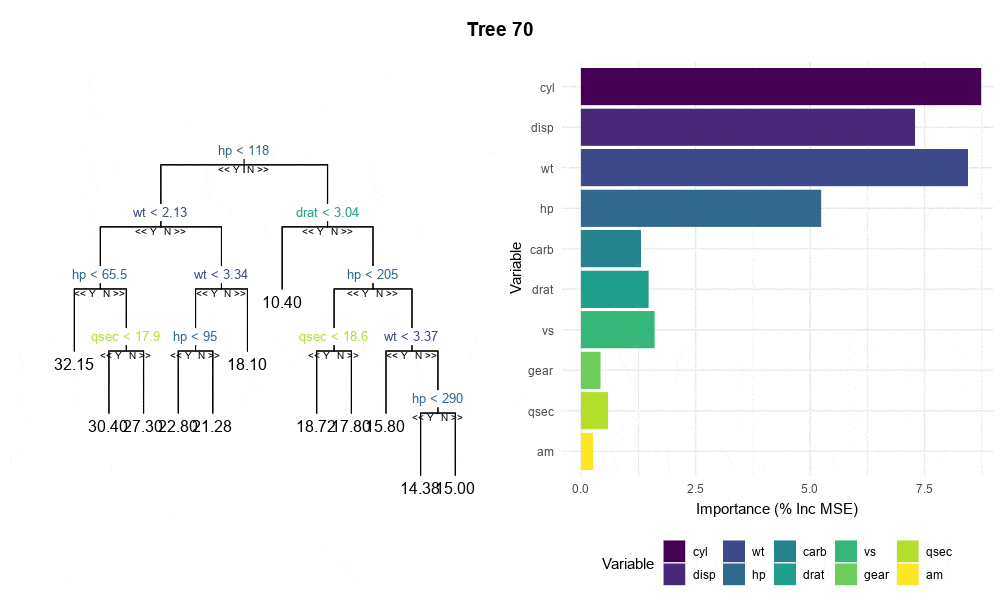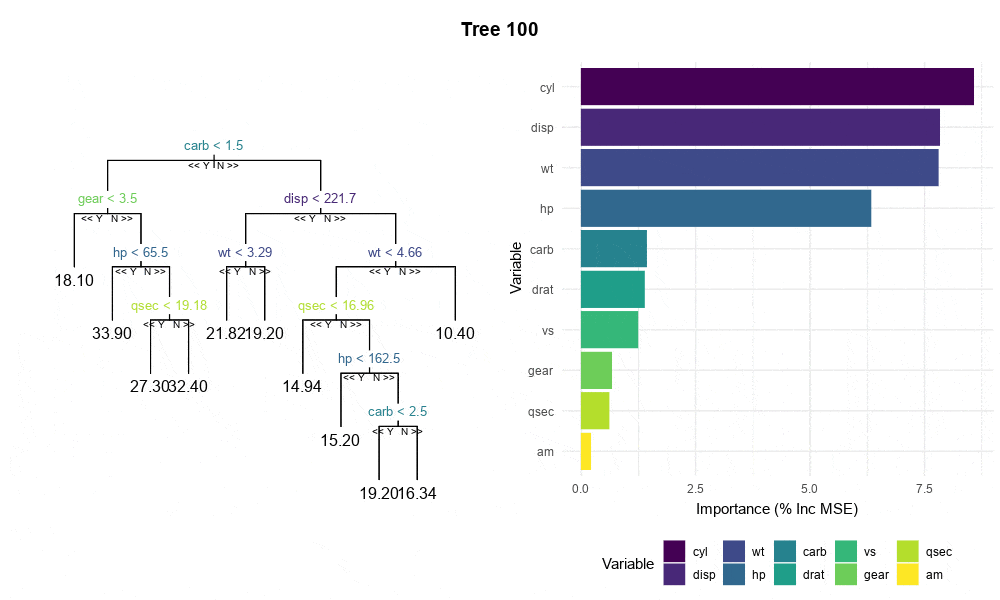
tree或rpart是否接受由randomForest构建的树,如果是,则可以使用这些软件包中的变量重要性函数。 - lmo