我正在寻找有效的方法来去除我的数据中的异常值。我尝试了一些在StackOverflow和其他地方找到的解决方案,但是它们都对我没有起作用(在1993年6月、1994年8月和1995年3月的样本数据中,应该检测到并删除4个高值,分别为21637、19590、21659和200000)。非常感谢任何建议!
以下是我目前测试过的内容:
数据概述如何从数据集中删除异常值
类似于第一种方法的问题
以下是我目前测试过的内容:
数据概述

仍然存在3个异常值,并且在时间序列的末尾删除了许多合法的高值。
y <- dat$Value
y_filter <- y[!y %in% boxplot.stats(y)$out]
plot(y_filter)

类似于第一种方法的问题
FindOutliers <- function(data) {
data <- data[!is.na(data)]
lowerq = quantile(data)[2]
upperq = quantile(data)[4]
iqr = upperq - lowerq #Or use IQR(data)
# we identify extreme outliers
extreme.threshold.upper = (iqr * 1.5) + upperq
extreme.threshold.lower = lowerq - (iqr * 1.5)
result <- which(data > extreme.threshold.upper | data < extreme.threshold.lower)
return(result)
}
# use the function to identify outliers
temp <- FindOutliers(y)
# remove the outliers
y1 <- y[-temp]
# show the data with the outliers removed
y1
#> [1] 511 524 310 721 514 318 511 511 318 510 21637 0
#> [13] 319 511 305 317 0 0 0 0 0 0 0 0
#> [25] 19590 0 0 0 0 0 0 0 0 21659 0 0
#> [37] 0 0 19 7 0 5 9 21 50 187 291 321
#> [49] 445 462 462 695 694 693 1276 2560 5500 11663 24307 25205
#> [61] 21667 18610 16739 14294 13517 12296 11247 11209 10954 11228 11387 11190
#> [73] 11193 11562 12279 11994 12073 11965 11386 10512 10677 10115 10118 9530
#> [85] 9016 9086 8167 8171 7561 7268 7359 7753 7168 7206 6926 6646
#> [97] 6674 6100 6177 5975 6033 5767 5497 5862 5594 5319
plot(y1)

library(performance)
outliers_list <- check_outliers(y) # Find outliers
outliers_list # Show the row index of the outliers
#> 9 outliers detected: cases 60, 61, 62, 63, 64, 65, 66, 67, 68.
#> - Based on the following method and threshold: zscore_robust (3.291).
#> - For variable: y.
#>
#> -----------------------------------------------------------------------------
#> Outliers per variable (zscore_robust):
#>
#> $y
#> Row Distance_Zscore_robust
#> 60 60 3.688817
#> 61 61 4.384524
#> 62 62 4.491276
#> 63 63 4.362517
#> 64 64 4.438994
#> 65 65 4.525319
#> 66 66 4.576871
#> 67 67 4.393886
#> 68 68 3.804809
str(outliers_list)
#> 'check_outliers' logi [1:116] FALSE FALSE FALSE FALSE FALSE FALSE ...
#> - attr(*, "data")='data.frame': 116 obs. of 4 variables:
#> ..$ Row : int [1:116] 1 2 3 4 5 6 7 8 9 10 ...
#> ..$ Distance_Zscore_robust: num [1:116] 0.645 0.643 0.669 0.619 0.644 ...
#> ..$ Outlier_Zscore_robust : num [1:116] 0 0 0 0 0 0 0 0 0 0 ...
#> ..$ Outlier : num [1:116] 0 0 0 0 0 0 0 0 0 0 ...
#> - attr(*, "threshold")=List of 1
#> ..$ zscore_robust: num 3.29
#> - attr(*, "method")= chr "zscore_robust"
#> - attr(*, "text_size")= num 3
#> - attr(*, "variables")= chr "y"
#> - attr(*, "raw_data")='data.frame': 116 obs. of 1 variable:
#> ..$ y: num [1:116] 511 524 310 721 514 318 511 511 318 510 ...
#> - attr(*, "outlier_var")=List of 1
#> ..$ zscore_robust:List of 1
#> .. ..$ y:'data.frame': 9 obs. of 2 variables:
#> .. .. ..$ Row : int [1:9] 60 61 62 63 64 65 66 67 68
#> .. .. ..$ Distance_Zscore_robust: num [1:9] 3.69 4.38 4.49 4.36 4.44 ...
#> - attr(*, "outlier_count")=List of 2
#> ..$ zscore_robust:'data.frame': 0 obs. of 2 variables:
#> .. ..$ Row : int(0)
#> .. ..$ n_Zscore_robust: int(0)
#> ..$ all :'data.frame': 0 obs. of 2 variables:
#> .. ..$ Row : num(0)
#> .. ..$ n_Zscore_robust: num(0)
y[!outliers_list]
#> [1] 511 524 310 721 514 318 511 511 318 510 21637 0
#> [13] 319 511 305 317 0 0 0 0 0 0 0 0
#> [25] 19590 0 0 0 0 0 0 0 0 21659 0 0
#> [37] 0 0 19 7 0 5 9 21 50 187 291 321
#> [49] 445 462 462 695 694 693 1276 2560 5500 11663 24307 30864
#> [61] 25205 21667 18610 16739 14294 13517 12296 11247 11209 10954 11228 11387
#> [73] 11190 11193 11562 12279 11994 12073 11965 11386 10512 10677 10115 10118
#> [85] 9530 9016 9086 8167 8171 7561 7268 7359 7753 7168 7206 6926
#> [97] 6646 6674 6100 6177 5975 6033 5767 5497 5862 5594 5319
par(mfrow = c(2,1), oma = c(2,2,0,0) + 0.1, mar = c(0,0,2,1) + 0.2)
plot(y)
plot(y[!outliers_list])

测试数据
library(tibble)
dat <- tibble::tribble(
~DateTime, ~Value,
"1993-06-06 11:00:00", NA,
"1993-06-06 12:00:00", 524,
"1993-06-06 13:00:00", 310,
"1993-06-06 14:00:00", 721,
"1993-06-06 15:00:00", 514,
"1993-06-06 16:00:00", 318,
"1993-06-06 17:00:00", 511,
"1993-06-06 18:00:00", 511,
"1993-06-06 19:00:00", 318,
"1993-06-06 20:00:00", 510,
"1993-06-06 21:00:00", 21637,
"1993-06-06 22:00:00", NA,
"1993-06-06 23:00:00", 319,
"1993-06-07 24:00:00", 511,
"1993-06-07 01:00:00", 305,
"1993-06-07 02:00:00", 317,
"1994-08-25 06:00:00", 0,
"1994-08-25 07:00:00", 0,
"1994-08-25 08:00:00", 0,
"1994-08-25 09:00:00", NA,
"1994-08-25 10:00:00", 0,
"1994-08-25 11:00:00", 0,
"1994-08-25 12:00:00", 0,
"1994-08-25 13:00:00", 0,
"1994-08-25 14:00:00", 19590,
"1994-08-26 06:00:00", 0,
"1994-08-26 07:00:00", 0,
"1994-08-26 08:00:00", 0,
"1994-08-26 09:00:00", 0,
"1994-08-26 10:00:00", NA,
"1994-08-26 11:00:00", NA,
"1994-08-26 12:00:00", 0,
"1994-08-26 13:00:00", 0,
"1994-08-26 14:00:00", 21659,
"1994-08-26 15:00:00", 0,
"1994-08-26 16:00:00", 0,
"1994-08-26 17:00:00", 0,
"1994-08-26 20:00:00", 0,
"1994-08-26 21:00:00", 19,
"1994-08-26 22:00:00", NA,
"1995-03-09 18:00:00", NA,
"1995-03-09 19:00:00", NA,
"1995-03-09 20:00:00", 9,
"1995-03-09 21:00:00", 21,
"1995-03-09 22:00:00", 50,
"1995-03-09 23:00:00", 187,
"1995-03-10 24:00:00", 291,
"1995-03-10 01:00:00", 321,
"1995-03-10 02:00:00", 445,
"1995-03-10 03:00:00", 2e+05,
"1995-03-10 04:00:00", 462,
"1995-03-10 05:00:00", 695,
"1995-03-10 06:00:00", 694,
"1995-03-10 07:00:00", 693,
"1995-03-10 08:00:00", 1276,
"1995-03-10 09:00:00", 2560,
"1995-03-10 10:00:00", 5500,
"1995-03-10 11:00:00", 11663,
"1995-03-10 12:00:00", 24307,
"1995-03-10 15:00:00", 36154,
"1995-03-10 17:00:00", 41876,
"1995-03-10 18:00:00", 42754,
"1995-03-10 19:00:00", NA,
"1995-03-10 20:00:00", NA,
"1995-03-10 21:00:00", 43034,
"1995-03-10 22:00:00", 43458,
"1995-03-10 23:00:00", 41953,
"1995-03-11 24:00:00", 37108,
"1995-03-11 01:00:00", 30864,
"1995-03-11 02:00:00", 25205,
"1995-03-11 03:00:00", NA,
"1995-03-11 04:00:00", NA,
"1995-03-11 05:00:00", NA,
"1995-03-11 06:00:00", NA,
"1995-03-11 07:00:00", 13517,
"1995-03-11 08:00:00", 12296,
"1995-03-11 09:00:00", 11247,
"1995-03-11 10:00:00", 11209,
"1995-03-11 11:00:00", 10954,
"1995-03-11 12:00:00", 11228,
"1995-03-11 13:00:00", 11387,
"1995-03-11 14:00:00", 11190,
"1995-03-11 15:00:00", 11193,
"1995-03-11 16:00:00", 11562,
"1995-03-11 17:00:00", 12279,
"1995-03-11 18:00:00", 11994,
"1995-03-11 19:00:00", 12073,
"1995-03-11 20:00:00", 11965,
"1995-03-11 21:00:00", 11386,
"1995-03-11 22:00:00", 10512,
"1995-03-11 23:00:00", 10677,
"1995-03-12 24:00:00", 10115,
"1995-03-12 01:00:00", 10118,
"1995-03-12 02:00:00", 9530,
"1995-03-12 03:00:00", 9016,
"1995-03-12 04:00:00", 9086,
"1995-03-12 05:00:00", 8167,
"1995-03-12 06:00:00", 8171,
"1995-03-12 07:00:00", 7561,
"1995-03-12 08:00:00", 7268,
"1995-03-12 09:00:00", 7359,
"1995-03-12 10:00:00", 7753,
"1995-03-12 11:00:00", 7168,
"1995-03-12 12:00:00", 7206,
"1995-03-12 13:00:00", 6926,
"1995-03-12 14:00:00", 6646,
"1995-03-12 15:00:00", 6674,
"1995-03-12 16:00:00", 6100,
"1995-03-12 17:00:00", 6177,
"1995-03-12 18:00:00", 5975,
"1995-03-12 19:00:00", 6033,
"1995-03-12 20:00:00", 5767,
"1995-03-12 21:00:00", 5497,
"1995-03-12 22:00:00", 5862,
"1995-03-12 23:00:00", 5594,
"1995-03-13 24:00:00", NA
)
2023-10-05创建,使用reprex v2.0.2
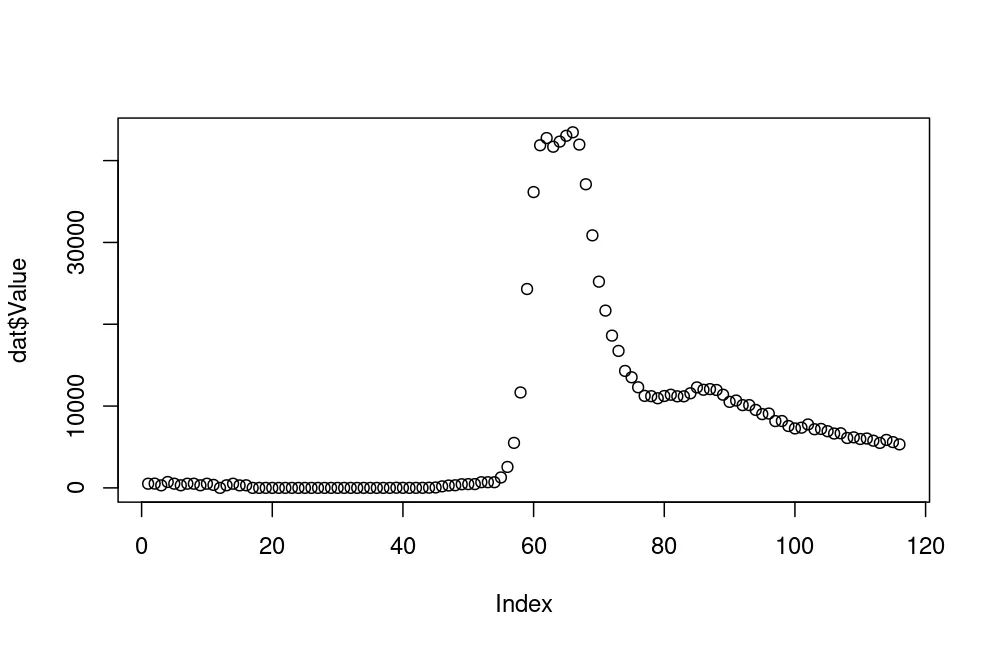


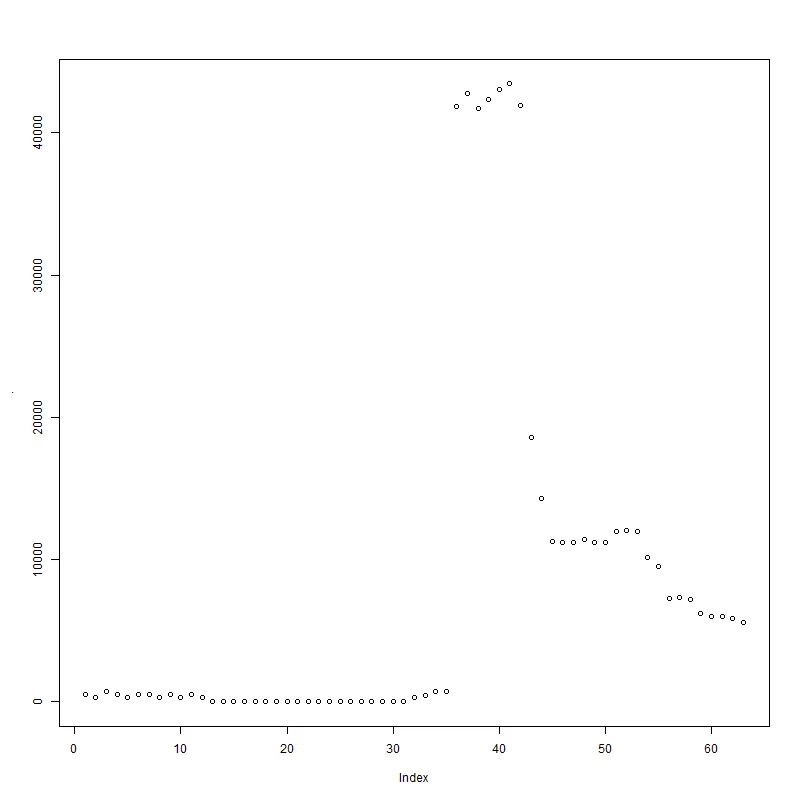
sp <- smooth.spline(dat$Value); dev <- abs(dat$Value - sp$y); dat$Value[dev > quantile(dev, 0.975)]看起来给出了一些有意义的结果? - undefined