在Python中,scipy.ndimage.morphology模块提供了distance_transform_edt函数。我将其应用于一个简单的案例,计算掩膜numpy数组中单个单元格到其他非null值单元格的欧几里得距离。
但是该函数会删除数组的掩膜,并按预期为每个具有非零值的单元格计算与空值参考单元格之间的欧几里得距离。
下面是我在我的博客文章中给出的示例:
%pylab
from scipy.ndimage.morphology import distance_transform_edt
l = 100
x, y = np.indices((l, l))
center1 = (50, 20)
center2 = (28, 24)
center3 = (30, 50)
center4 = (60,48)
radius1, radius2, radius3, radius4 = 15, 12, 19, 12
circle1 = (x - center1[0])**2 + (y - center1[1])**2 < radius1**2
circle2 = (x - center2[0])**2 + (y - center2[1])**2 < radius2**2
circle3 = (x - center3[0])**2 + (y - center3[1])**2 < radius3**2
circle4 = (x - center4[0])**2 + (y - center4[1])**2 < radius4**2
# 3 circles
img = circle1 + circle2 + circle3 + circle4
mask = ~img.astype(bool)
img = img.astype(float)
m = ones_like(img)
m[center1] = 0
#imshow(distance_transform_edt(m), interpolation='nearest')
m = ma.masked_array(distance_transform_edt(m), mask)
imshow(m, interpolation='nearest')
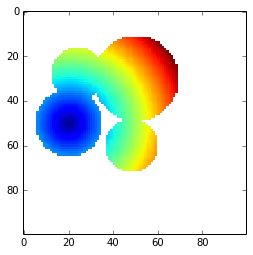
然而,我想计算考虑数组中掩码元素的测地线距离变换。我不想计算通过掩码元素的直线欧几里得距离。
我使用了 Dijkstra 算法来获得我想要的结果。下面是我提出的实现:
def geodesic_distance_transform(m):
mask = m.mask
visit_mask = mask.copy() # mask visited cells
m = m.filled(numpy.inf)
m[m!=0] = numpy.inf
distance_increments = numpy.asarray([sqrt(2), 1., sqrt(2), 1., 1., sqrt(2), 1., sqrt(2)])
connectivity = [(i,j) for i in [-1, 0, 1] for j in [-1, 0, 1] if (not (i == j == 0))]
cc = unravel_index(m.argmin(), m.shape) # current_cell
while (~visit_mask).sum() > 0:
neighbors = [tuple(e) for e in asarray(cc) - connectivity
if not visit_mask[tuple(e)]]
tentative_distance = [distance_increments[i] for i,e in enumerate(asarray(cc) - connectivity)
if not visit_mask[tuple(e)]]
for i,e in enumerate(neighbors):
d = tentative_distance[i] + m[cc]
if d < m[e]:
m[e] = d
visit_mask[cc] = True
m_mask = ma.masked_array(m, visit_mask)
cc = unravel_index(m_mask.argmin(), m.shape)
return m
gdt = geodesic_distance_transform(m)
imshow(gdt, interpolation='nearest')
colorbar()
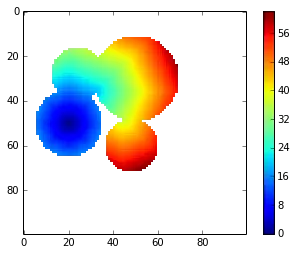
上面实现的函数可以正常工作,但对于我开发的应用程序来说太慢了,需要多次计算测地距离变换。
下面是欧几里得距离变换和测地距离变换的时间基准:
%timeit distance_transform_edt(m)
1000 loops, best of 3: 1.07 ms per loop
%timeit geodesic_distance_transform(m)
1 loops, best of 3: 702 ms per loop
如何获得更快的测地线距离转换?
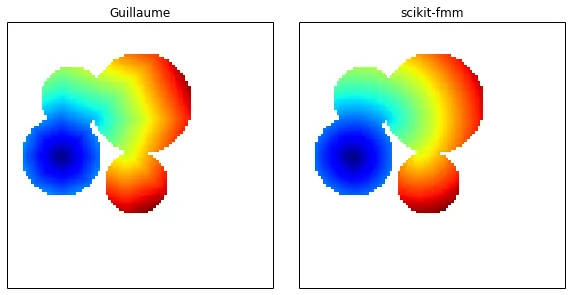

distance_transform_edt并不能给出你想要的结果,但是在将其输入函数之前,你的m也从未被掩蔽。 - Jason