4个回答
36
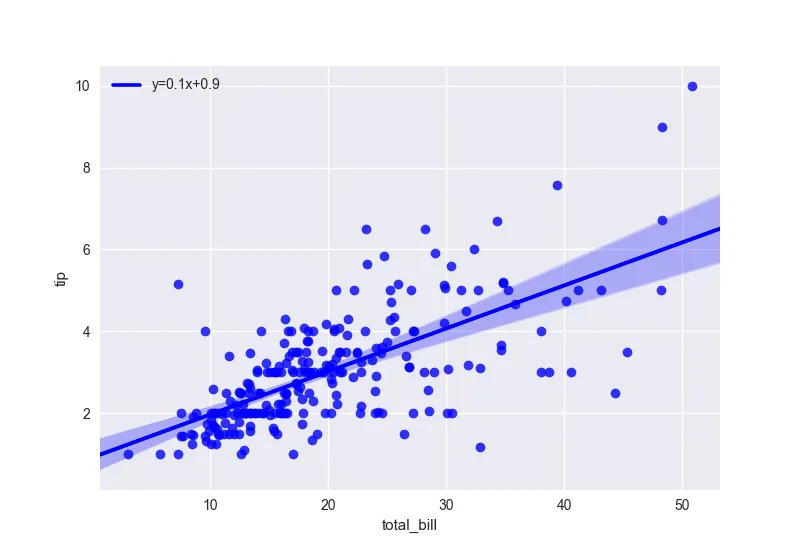
您可以使用线性拟合系数来制作类似于此示例中的图例:
import seaborn as sns
import matplotlib.pyplot as plt
from scipy import stats
tips = sns.load_dataset("tips")
# get coeffs of linear fit
slope, intercept, r_value, p_value, std_err = stats.linregress(tips['total_bill'],tips['tip'])
# use line_kws to set line label for legend
ax = sns.regplot(x="total_bill", y="tip", data=tips, color='b',
line_kws={'label':"y={0:.1f}x+{1:.1f}".format(slope,intercept)})
# plot legend
ax.legend()
plt.show()
如果你使用更复杂的拟合函数,你可以使用 LaTeX 公式:https://matplotlib.org/users/usetex.html
- Serenity
2
14您想知道如何确定regplot线反映了scipy.stats回归参数?令人惊讶的是,Seaborn并未提供用于制作其图形的计算参数... - joaquin
1@joaquin,不幸的是他们也没有计划这样做。https://dev59.com/bFwX5IYBdhLWcg3wtBQa#47062135 - Abdelrahman Shoman
6
如果使用 seaborn 的 lmplot,要注释多个线性回归线,可以采取以下方法。
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
df = pd.read_excel('data.xlsx')
# assume some random columns called EAV and PAV in your DataFrame
# assume a third variable used for grouping called "Mammal" which will be used for color coding
p = sns.lmplot(x=EAV, y=PAV,
data=df, hue='Mammal',
line_kws={'label':"Linear Reg"}, legend=True)
ax = p.axes[0, 0]
ax.legend()
leg = ax.get_legend()
L_labels = leg.get_texts()
# assuming you computed r_squared which is the coefficient of determination somewhere else
slope, intercept, r_value, p_value, std_err = stats.linregress(df['EAV'],df['PAV'])
label_line_1 = r'$y={0:.1f}x+{1:.1f}'.format(slope,intercept)
label_line_2 = r'$R^2:{0:.2f}$'.format(0.21) # as an exampple or whatever you want[!
L_labels[0].set_text(label_line_1)
L_labels[1].set_text(label_line_2)
结果:
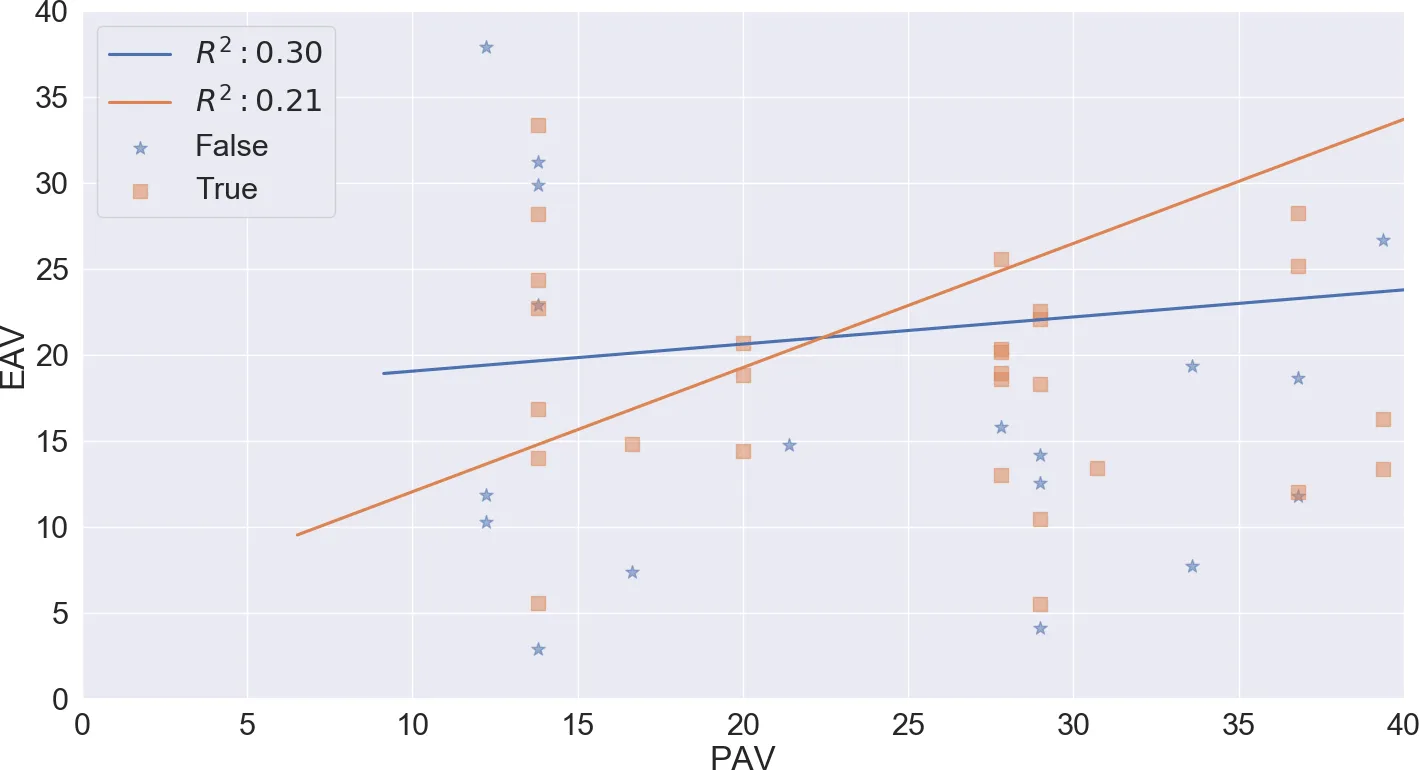
- RMS
2
我不明白为什么你在label_line_2中放置0.21(“或者任何你想要的”,用你的话来说),而不是使用stats.linregress提供的实际r_value ** 2。 - Robyc
这是一个例子,当然你可以做到。问题是要注释线性回归线,而不是如何在注释中写入R平方值。 - RMS
2
更简单的语法,同样的结果。
import seaborn as sns
import matplotlib.pyplot as plt
from scipy import stats
slope, intercept, r_value, pv, se = stats.linregress(df['alcohol'],df['magnesium'])
sns.regplot(x="alcohol", y="magnesium", data=df,
ci=None, label="y={0:.1f}x+{1:.1f}".format(slope, intercept)).legend(loc="best")
- Union find
1
当然,您也可以在Python3中使用f字符串。 - Union find
1
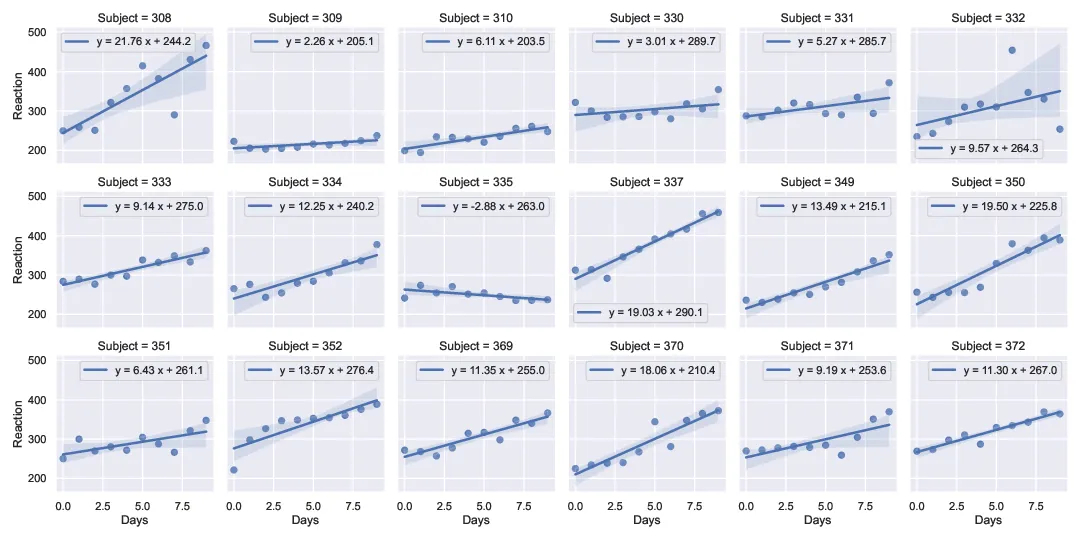
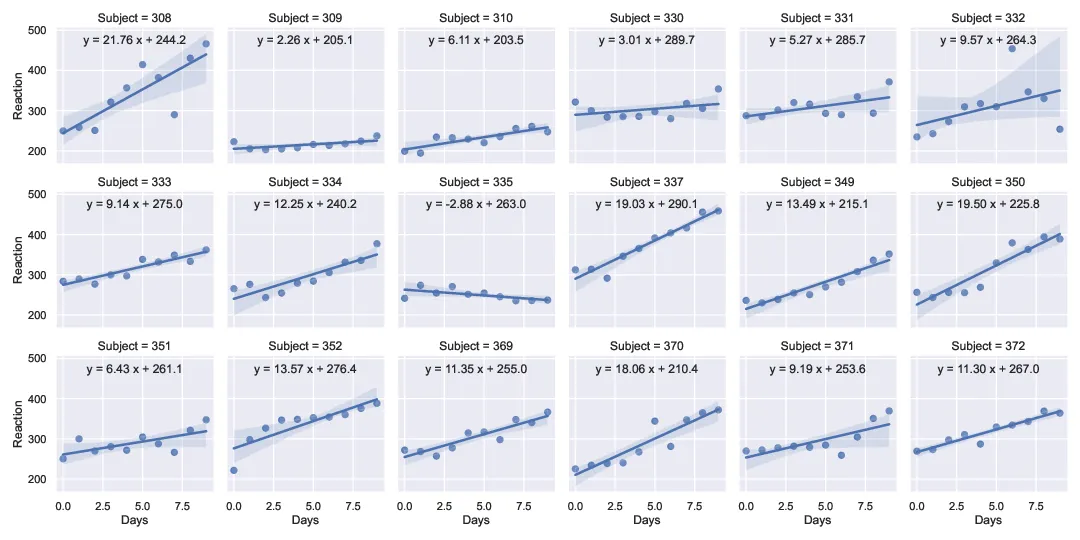
我扩展了@RMS的解决方案,使其适用于多面板lmplot示例(使用睡眠剥夺研究(Belenky等人,J Sleep Res 2003)中的数据,在pydataset中可用)。这样可以在不使用例如regplot和plt.subplots的情况下具有轴特定的图例/标签。
编辑:根据这里Marcos的答案建议,添加了使用FacetGrid()的map_dataframe()方法的第二种方法。
import numpy as np
import scipy as sp
import pandas as pd
import seaborn as sns
import pydataset as pds
import matplotlib.pyplot as plt
# use seaborn theme
sns.set_theme(color_codes=True)
# Load data from sleep deprivation study (Belenky et al, J Sleep Res 2003)
# ['Reaction', 'Days', 'Subject'] = [reaction time (ms), deprivation time, Subj. No.]
df = pds.data("sleepstudy")
# convert integer label to string
df['Subject'] = df['Subject'].apply(str)
# perform linear regressions outside of seaborn to get parameters
subjects = np.unique(df['Subject'].to_numpy())
fit_str = []
for s in subjects:
ddf = df[df['Subject'] == s]
m, b, r_value, p_value, std_err = \
sp.stats.linregress(ddf['Days'],ddf['Reaction'])
fs = f"y = {m:.2f} x + {b:.1f}"
fit_str.append(fs)
method_one = False
method_two = True
if method_one:
# Access legend on each axis to write equation
#
# Create 18 panel lmplot with seaborn
g = sns.lmplot(x="Days", y="Reaction", col="Subject",
col_wrap=6, height=2.5, data=df,
line_kws={'label':"Linear Reg"}, legend=True)
# write string with fit result into legend string of each axis
axes = g.axes # 18 element list of axes objects
i=0
for ax in axes:
ax.legend() # create legend on axis
leg = ax.get_legend()
leg_labels = leg.get_texts()
leg_labels[0].set_text(fit_str[i])
i += 1
elif method_two:
# use the .map_dataframe () method from FacetGrid() to annotate plot
# https://dev59.com/cl8e5IYBdhLWcg3wlbTx (answer by @Marcos)
#
# Create 18 panel lmplot with seaborn
g = sns.lmplot(x="Days", y="Reaction", col="Subject",
col_wrap=6, height=2.5, data=df)
def annotate(data, **kws):
m, b, r_value, p_value, std_err = \
sp.stats.linregress(data['Days'],data['Reaction'])
ax = plt.gca()
ax.text(0.5, 0.9, f"y = {m:.2f} x + {b:.1f}",
horizontalalignment='center',
verticalalignment='center',
transform=ax.transAxes)
g.map_dataframe(annotate)
# write figure to pdf
plt.savefig("sleepstudy_data_w-fits.pdf")
更新于2022-05-11:与绘图技术无关,事实证明这些数据的解释是错误的(例如在原始R存储库中提供的解释)。请参见此处报告的问题。适合进行的拟合应该是从第2到第9天,对应于零到七天的睡眠剥夺(每晚3小时睡眠)。前三个数据点对应于训练和基线天数(每晚8小时睡眠)。
- Ben H
网页内容由stack overflow 提供, 点击上面的可以查看英文原文,
原文链接
原文链接