no,store_id,revenue,profit,state,country
0,101,779183,281257,WD,India
1,101,144829,838451,WD,India
2,101,766465,757565,AL,Japan
我的代码如下
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
df1 = pd.get_dummies(df, columns=['state','country'])
clusters = 2
km = KMeans(n_clusters=8).fit(df1)
labels = km.predict(df1)
df1['cluster_id'] = km.labels_
def distance_to_centroid(row, centroid):
row = row[['no','store_id','revenue','profit','state','country']]
return euclidean(row, centroid)
df1['distance_to_center0'] = df1.apply(lambda r: distance_to_centroid(r,
km.cluster_centers_[0]),1)
df1['distance_to_center1'] = df1.apply(lambda r: distance_to_centroid(r,
km.cluster_centers_[1]),1)
dummies_df =dummies[['distance_to_center0','distance_to_center1','cluster_id']]
test = {0:"Blue", 1:"Red", 2:"Green",3:"Black",4:"Orange",5:"Yellow",6:"Violet",7:"Grey"}
sns.scatterplot(x="distance_to_center0", y="distance_to_center1", data=dummies_df, hue="cluster_id", palette = test)
为了找到中心点,下面是代码。
km = KMeans(n_clusters=7).fit(dummies)
closest, _ = pairwise_distances_argmin_min(km.cluster_centers_, dummies)
closest
如何为聚类绘制散点图
如何将打印点与聚类分开
就像最小异常值方法中-1是离群值(scikit learn)。kmeans.labes_仅打印1和0,如何获取离群值
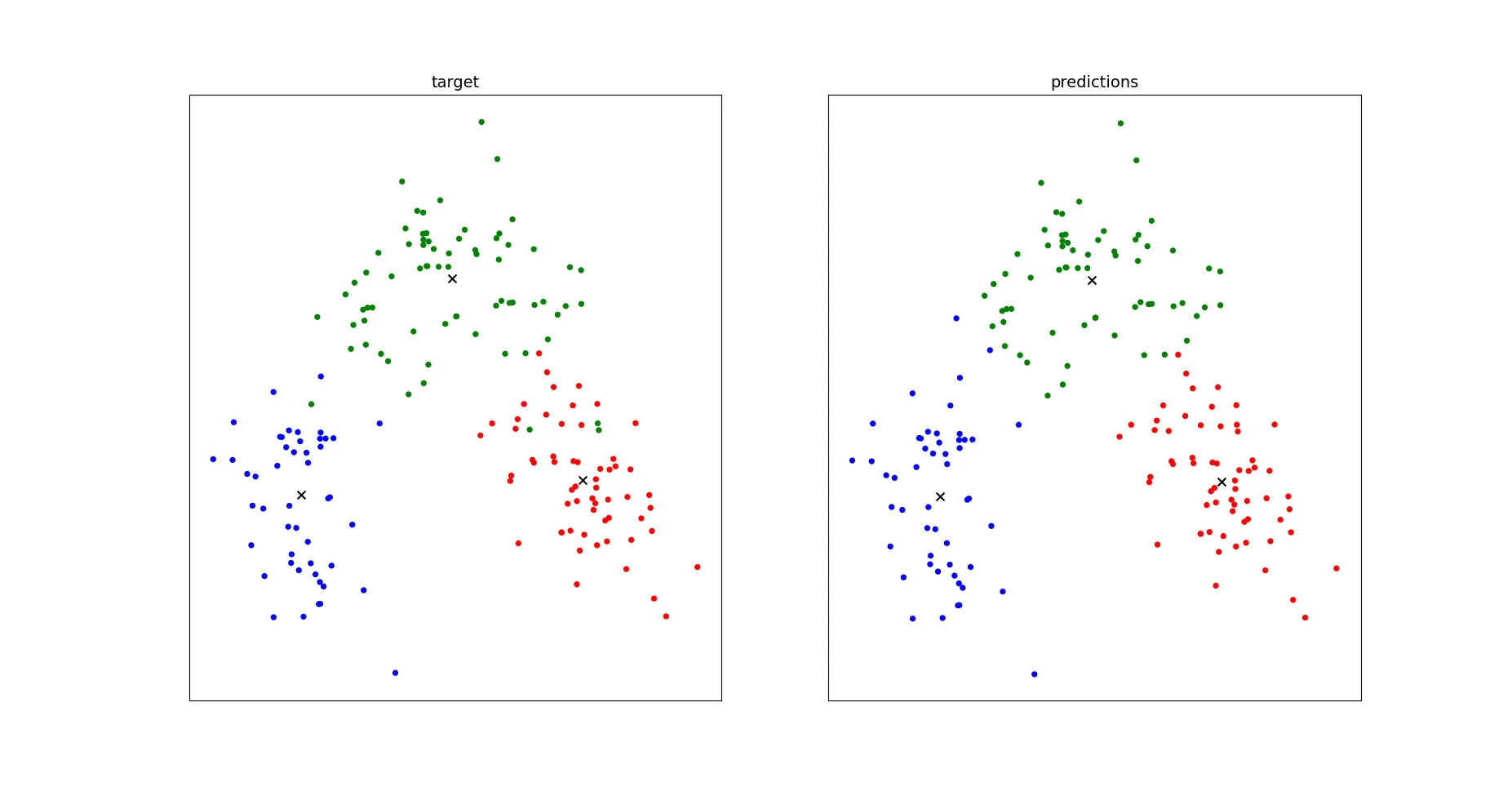
plt.scatter(x, y)。你所说的“将打印点远离聚类”是什么意思? - moe asal