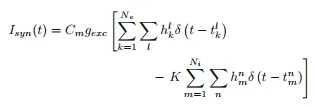我正在处理的模型有一个神经元(由Hodgkin-Huxley方程建模),这个神经元本身接收来自其他神经元的一堆突触输入,因为它处于网络中。建模输入的标准方法是使用由一堆Δ函数脉冲组成的尖峰列,以指定速率到达,作为泊松过程。其中一些脉冲会对神经元产生兴奋反应,而另一些则提供抑制性脉冲。因此,突触电流应该如下所示:
在这里,Ne是兴奋性神经元的数量,Ni是抑制性神经元的数量,h要么是0要么是1(以概率p为1),表示是否成功传递了一个脉冲,而Δ函数中的tkl是第k个神经元的第l次脉冲放电时间($tm^n$也是如此)。因此,我们尝试编写代码的基本思想是首先假设我有100个神经元向我的HH神经元提供脉冲(其中80个是兴奋性的,20个是抑制性的)。然后,我们形成了一个数组,其中一列枚举神经元(使神经元#0-79是兴奋性神经元,#80-99是抑制性神经元)。然后,我们检查某个时间间隔内是否有脉冲,如果有,选择0-1之间的随机数,如果它小于指定的概率p,则将其分配为1,否则将其设置为0。然后,我们绘制电压随时间变化的图形,以查看神经元何时发生脉冲。
我认为代码是可行的,但问题在于,一旦我在网络中添加了更多的神经元(一篇论文声称他们使用了5000个神经元),运行时间就会非常长,这对于进行数值模拟来说是不可行的。我的问题是:是否有更好的方法来模拟脉冲输入到神经元中,以便在网络中有大量神经元时计算速度更快?以下是我们尝试过的代码(它有点长,因为HH方程非常详细):
import scipy as sp
import numpy as np
import pylab as plt
#Constants
C_m = 1.0 #membrane capacitance, in uF/cm^2"""
g_Na = 120.0 #Sodium (Na) maximum conductances, in mS/cm^2""
g_K = 36.0 #Postassium (K) maximum conductances, in mS/cm^2"""
g_L = 0.3 #Leak maximum conductances, in mS/cm^2"""
E_Na = 50.0 #Sodium (Na) Nernst reversal potentials, in mV"""
E_K = -77.0 #Postassium (K) Nernst reversal potentials, in mV"""
E_L = -54.387 #Leak Nernst reversal potentials, in mV"""
def poisson_spikes(t, N=100, rate=1.0 ):
spks = []
dt = t[1] - t[0]
for n in range(N):
spkt = t[np.random.rand(len(t)) < rate*dt/1000.] #Determine list of times of spikes
idx = [n]*len(spkt) #Create vector for neuron ID number the same length as time
spkn = np.concatenate([[idx], [spkt]], axis=0).T #Combine tw lists
if len(spkn)>0:
spks.append(spkn)
spks = np.concatenate(spks, axis=0)
return spks
N = 100
N_ex = 80 #(0..79)
N_in = 20 #(80..99)
G_ex = 1.0
K = 4
dt = 0.01
t = sp.arange(0.0, 300.0, dt) #The time to integrate over """
ic = [-65, 0.05, 0.6, 0.32]
spks = poisson_spikes(t, N, rate=10.)
def alpha_m(V):
return 0.1*(V+40.0)/(1.0 - sp.exp(-(V+40.0) / 10.0))
def beta_m(V):
return 4.0*sp.exp(-(V+65.0) / 18.0)
def alpha_h(V):
return 0.07*sp.exp(-(V+65.0) / 20.0)
def beta_h(V):
return 1.0/(1.0 + sp.exp(-(V+35.0) / 10.0))
def alpha_n(V):
return 0.01*(V+55.0)/(1.0 - sp.exp(-(V+55.0) / 10.0))
def beta_n(V):
return 0.125*sp.exp(-(V+65) / 80.0)
def I_Na(V, m, h):
return g_Na * m**3 * h * (V - E_Na)
def I_K(V, n):
return g_K * n**4 * (V - E_K)
def I_L(V):
return g_L * (V - E_L)
def I_app(t):
return 3
def I_syn(spks, t):
"""
Synaptic current
spks = [[synid, t],]
"""
exspk = spks[spks[:,0]<N_ex] # Check for all excitatory spikes
delta_k = exspk[:,1] == t # Delta function
if sum(delta_k) > 0:
h_k = np.random.rand(len(delta_k)) < 0.5 # p = 0.5
else:
h_k = 0
inspk = spks[spks[:,0] >= N_ex] #Check remaining neurons for inhibitory spikes
delta_m = inspk[:,1] == t #Delta function for inhibitory neurons
if sum(delta_m) > 0:
h_m = np.random.rand(len(delta_m)) < 0.5 #p =0.5
else:
h_m = 0
isyn = C_m*G_ex*(np.sum(h_k*delta_k) - K*np.sum(h_m*delta_m))
return isyn
def dALLdt(X, t):
V, m, h, n = X
dVdt = (I_app(t)+I_syn(spks,t)-I_Na(V, m, h) - I_K(V, n) - I_L(V)) / C_m
dmdt = alpha_m(V)*(1.0-m) - beta_m(V)*m
dhdt = alpha_h(V)*(1.0-h) - beta_h(V)*h
dndt = alpha_n(V)*(1.0-n) - beta_n(V)*n
return np.array([dVdt, dmdt, dhdt, dndt])
X = [ic]
for i in t[1:]:
dx = (dALLdt(X[-1],i))
x = X[-1]+dt*dx
X.append(x)
X = np.array(X)
V = X[:,0]
m = X[:,1]
h = X[:,2]
n = X[:,3]
ina = I_Na(V, m, h)
ik = I_K(V, n)
il = I_L(V)
plt.figure()
plt.subplot(3,1,1)
plt.title('Hodgkin-Huxley Neuron')
plt.plot(t, V, 'k')
plt.ylabel('V (mV)')
plt.subplot(3,1,2)
plt.plot(t, ina, 'c', label='$I_{Na}$')
plt.plot(t, ik, 'y', label='$I_{K}$')
plt.plot(t, il, 'm', label='$I_{L}$')
plt.ylabel('Current')
plt.legend()
plt.subplot(3,1,3)
plt.plot(t, m, 'r', label='m')
plt.plot(t, h, 'g', label='h')
plt.plot(t, n, 'b', label='n')
plt.ylabel('Gating Value')
plt.legend()
plt.show()
我不熟悉其他专门用于神经网络的软件包,但我想编写自己的软件包,主要是因为我计划进行随机分析,这需要相当多的数学细节,而我不知道那些软件包是否提供这样的细节。