我正在进行图分析。 我想计算一个N乘N的相似性矩阵,其中包含每两个顶点之间的Adamic Adar相似度。 为了概述Adamic Adar,让我从这个介绍开始:给定一个无向图G的邻接矩阵A。CN是两个顶点x、y的所有公共邻居的集合。两个顶点的公共邻居是指两个顶点都有一条边/链接到该邻居节点,即在A中对应的公共邻居节点上,两个顶点都将具有1。kn是节点n的度数。
Adamic-Adar定义如下: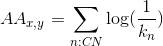 我的计算尝试是从A中提取x和y节点的行,然后将它们相加。 然后查找值为2的元素,然后获取它们的度数并应用方程式。 但是,计算非常耗费时间。 我尝试使用一个包含1032个顶点的图表进行计算,但需要很长时间才能计算。 它开始耗时7分钟,然后我取消了计算。 所以我的问题是:是否有更好的算法来计算?
我的计算尝试是从A中提取x和y节点的行,然后将它们相加。 然后查找值为2的元素,然后获取它们的度数并应用方程式。 但是,计算非常耗费时间。 我尝试使用一个包含1032个顶点的图表进行计算,但需要很长时间才能计算。 它开始耗时7分钟,然后我取消了计算。 所以我的问题是:是否有更好的算法来计算?
以下是我的python代码:
Adamic-Adar定义如下:
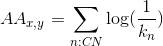 我的计算尝试是从A中提取x和y节点的行,然后将它们相加。 然后查找值为2的元素,然后获取它们的度数并应用方程式。 但是,计算非常耗费时间。 我尝试使用一个包含1032个顶点的图表进行计算,但需要很长时间才能计算。 它开始耗时7分钟,然后我取消了计算。 所以我的问题是:是否有更好的算法来计算?
我的计算尝试是从A中提取x和y节点的行,然后将它们相加。 然后查找值为2的元素,然后获取它们的度数并应用方程式。 但是,计算非常耗费时间。 我尝试使用一个包含1032个顶点的图表进行计算,但需要很长时间才能计算。 它开始耗时7分钟,然后我取消了计算。 所以我的问题是:是否有更好的算法来计算?以下是我的python代码:
def aa(graph):
"""
Calculates the Adamic-Adar index.
"""
N = graph.num_vertices()
A = gts.adjacency(graph)
S = np.zeros((N,N))
degrees = get_degrees_dic(graph)
for i in xrange(N):
A_i = A[i]
for j in xrange(N):
if j != i:
A_j = A[j]
intersection = A_i + A_j
common_ns_degs = list()
for index in xrange(N):
if intersection[index] == 2:
cn_deg = degrees[index]
common_ns_degs.append(1.0/np.log10(cn_deg))
S[i,j] = np.sum(common_ns_degs)
return S