背景
我正在尝试预测产品线的销售情况(在最后的样本中表示为y_test)。其时间段内的销售量基于另一个产品的所有先前销售情况(x_test),以及这些先前销售情况中有多少仍在使用。然而,直接测量先前销售的产品中有多少仍在使用是不可能的,因此需要推断出生存曲线。
例如,如果您制造特定智能手机型号的配件,那么配件销售量至少部分基于仍在使用的智能手机数量。(顺便说一下,这不是作业。)
细节
我有一些时间序列数据,并希望使用glm或类似方法来拟合回归模型。依赖变量和独立变量之间的关系如下:
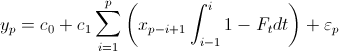
其中p是时间期,yp是依赖变量,xp是独立变量,c0和c1是回归系数,Ft是累积分布函数(例如pgamma),ep是残差项。
在前三个时间段内,该函数会扩展为以下形式:
#y[1] = c0 + c1*(x[1]*(1-integrate(function(q) {pgamma(q, c2, c2/c3)}, 0, 1)$value))
#y[2] = c0 + c1*(x[1]*(1-integrate(function(q) {pgamma(q, c2, c2/c3)}, 1, 2)$value) + x[2]*(1-integrate(function(q) {pgamma(q, c2, c2/c3)}, 0, 1)$value))
#y[3] = c0 + c1*(x[1]*(1-integrate(function(q) {pgamma(q, c2, c2/c3)}, 2, 3)$value) + x[2]*(1-integrate(function(q) {pgamma(q, c2, c2/c3)}, 1, 2)$value) + x[3]*(1-integrate(function(q) {pgamma(q, c2, c2/c3)}, 0, 1)$value))
因此,我有xp和yp的历史数据,并且想要获取最小化残差的系数/参数c0、c1、c2和c3的值。
我认为解决方案是使用glm并创建自定义family,但我不确定如何做到这一点。 我查看了Gamma family的代码,但没有取得太大进展。我已经能够使用nlminb手动进行优化,但我更喜欢glm或类似函数提供的简单性和有用性(即predict等)。
这里是一些示例数据:
# Survival function (the integral part):
fsurv<-function(q, par) {
l<-length(q)
out<-vapply(1:l, function(i) {1-integrate(function(x) {pgamma(x, par[1], par[1]/par[2])}, q[i]-1, q[i])$value}, FUN.VALUE=0)
return(out)}
# Sum up the products:
frevsumprod <- function(x,y) {
l <- length(y)
out <- vapply(1:l, function(i) sum(x[1:i]*rev(y[1:i])), FUN.VALUE=0)
return(out)}
# Sample data:
p<-1:24 # Number of periods
x_test<-c(1188, 2742, 4132) # Sample data
y_test<-c(82520, 308910, 749395, 801905, 852310, 713935, 624170, 603960, 640660, 553600, 497775, 444140) # Sample data
c<-c(-50.161147,128.787437,0.817085,13.845487) # Coefficients and parameters, from another method that fit the data
# Pad the data to the correct length:
pad<-function(p,v,padval=0) {
l<-length(p)
padv<-l-length(v)
if(padv>0) (v<-c(v,rep(padval,padv)))
return(v)
}
x_test<-pad(p,x_test)
y_test<-pad(p,y_test,NA)
y_fitted<-c[0+1]+c[1+1]*frevsumprod(x_test,fsurv(p,c[(2:3)+1])) # Fitted values from regression
library(ggplot2)
ggplot(data.frame(p,y_test,y_fitted))+geom_point(aes(p,y_test))+geom_line(aes(p,y_fitted)) # Plot actual and fit