1个回答
2
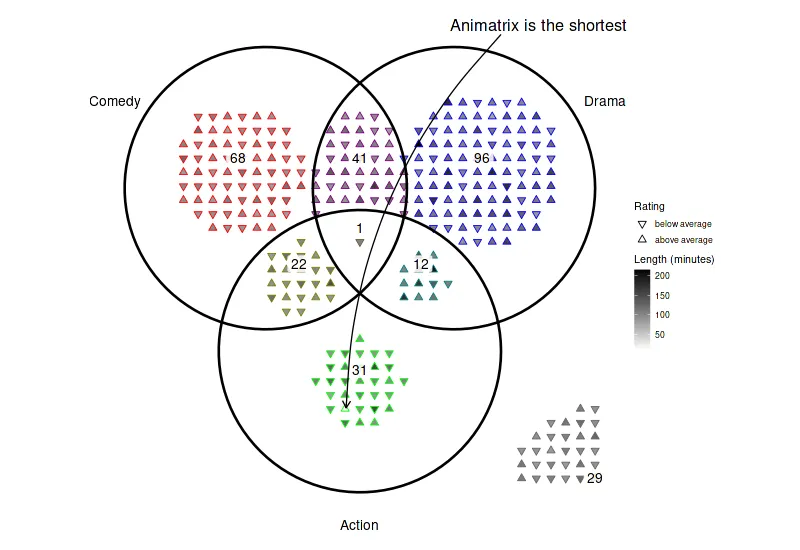
complex-upset提供了在R中创建点密度图的工具(遵循ggplot2方法,详见文档)。
让我们加载并过滤一些数据作为示例:
首先,您需要整理数据。
然后,你可以使用标准的ggplot2函数和由complex-upset提供的新几何对象进行绘图:
它还可以注释单个点(以及更多!) 但是这个实现有一些限制:
library(ggplot2)
library(ComplexUpset)
movies = as.data.frame(ggplot2movies::movies)
genres = c('Comedy', 'Drama', 'Action')
movies[genres] = movies[genres] == 1
movies[movies$mpaa == '', 'mpaa'] = NA
movies = na.omit(movies)
首先,您需要整理数据。
movies_subset = head(movies, 300)
movies_subset$good_rating = movies_subset$rating > mean(movies_subset$rating)
arranged = arrange_venn(movies_subset, sets=genres)
然后,你可以使用标准的ggplot2函数和由complex-upset提供的新几何对象进行绘图:
geom_venn_region、geom_venn_circle、geom_venn_label_set。
(
ggplot(arranged)
+ theme_void()
+ coord_fixed()
+ geom_venn_region(movies_subset, sets=genres_subset, alpha=0.2)
+ geom_point(aes(x=x, y=y, color=region), size=1.5)
+ geom_venn_circle(movies_subset, sets=genres_subset, size=2)
+ geom_venn_label_set(movies_subset, sets=genres_subset, aes(label=region), outwards_adjust=2.6)
+ scale_color_venn_mix(movies, sets=genres_subset, guide='none')
+ scale_fill_venn_mix(
movies, sets=genres_subset,
guide='none',
highlight=c('Comedy-Action', 'Drama'),
inactive_color='white'
)
)
它还可以注释单个点(以及更多!) 但是这个实现有一些限制:
- 它只支持最多三组
- 在大型数据集(>1000个观察)中进行排列速度较慢
- 通常需要微调排列参数才能获得所需的结果
- 截至撰写本文的当天,最新版本提供了一些次要改进,可以从GitHub安装,但不能从CRAN安装(尚未)。
- krassowski
网页内容由stack overflow 提供, 点击上面的可以查看英文原文,
原文链接
原文链接