made4的heatplot,另一个是使用heatmap.2中的gplots。适当的结果取决于分析,但我正在尝试理解为什么它们的默认值如此之不同,以及如何使这两个函数给出相同(或类似)的结果,以便我可以理解进入其中的所有“黑匣子”参数。以下是示例数据和包:
require(gplots)
# made4 from bioconductor
require(made4)
data(khan)
data <- as.matrix(khan$train[1:30,])
使用heatmap.2对数据进行聚类,得到如下结果:
heatmap.2(data, trace="none")
使用
heatplot 会得到以下结果:heatplot(data)
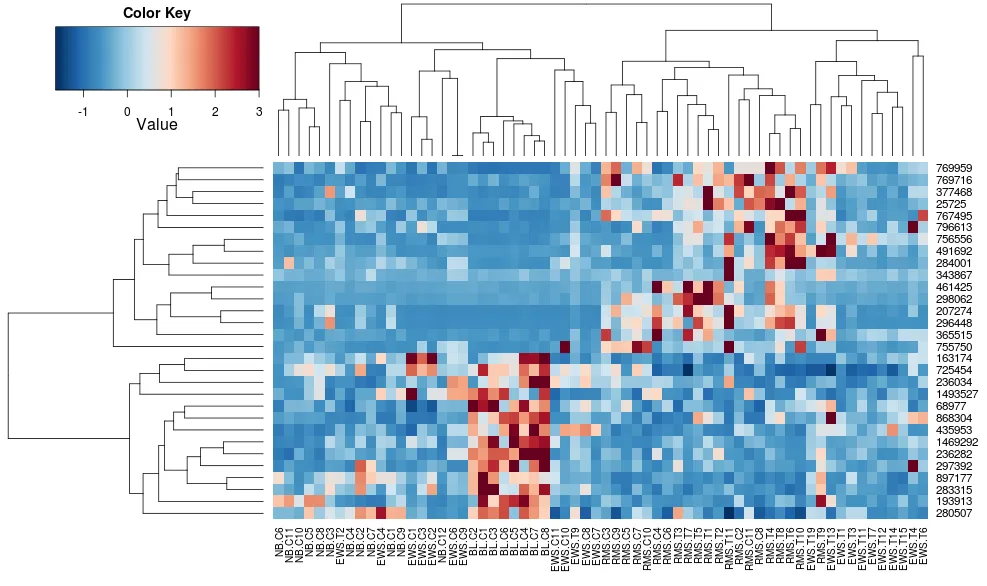
最初的结果和比例尺非常不同。在这种情况下,heatplot 的结果看起来更合理,因此我想了解应该将哪些参数输入到 heatmap.2 中,以使其执行相同的操作,因为 heatmap.2 具有其他优点/功能,我想使用它,并且因为我想了解缺少的要素。
heatplot 使用相关距离的平均连接,因此我们可以将其输入到 heatmap.2 中,以确保使用类似的聚类(基于:https://stat.ethz.ch/pipermail/bioconductor/2010-August/034757.html)。
dist.pear <- function(x) as.dist(1-cor(t(x)))
hclust.ave <- function(x) hclust(x, method="average")
heatmap.2(data, trace="none", distfun=dist.pear, hclustfun=hclust.ave)
导致结果如下:
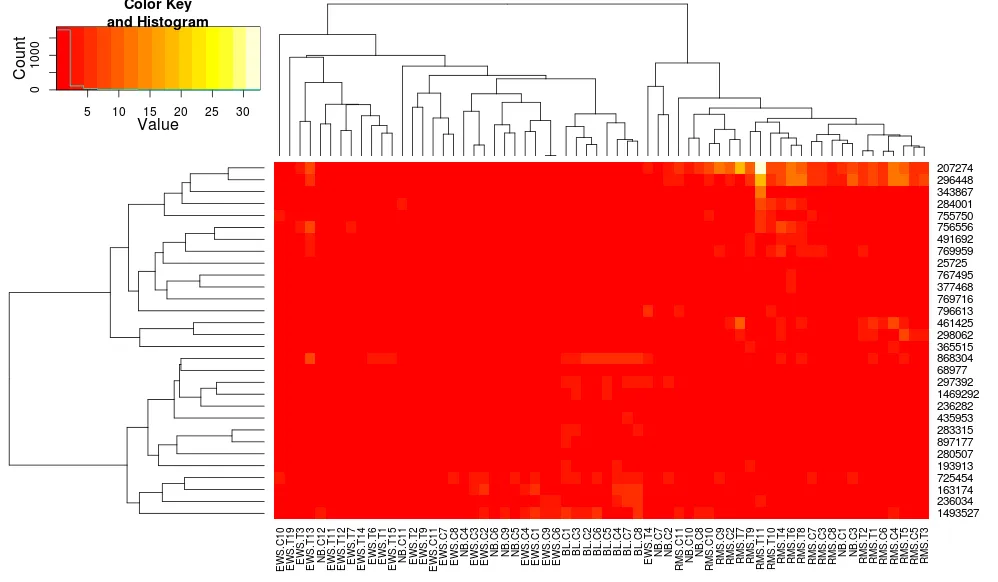 这使得行侧树状图看起来更相似,但列仍然不同,比例也不同。看起来
这使得行侧树状图看起来更相似,但列仍然不同,比例也不同。看起来heatplot默认情况下以某种方式缩放列,而heatmap.2默认情况下不会这样做。如果我在heatmap.2中添加行缩放,我会得到:heatmap.2(data, trace="none", distfun=dist.pear, hclustfun=hclust.ave,scale="row")
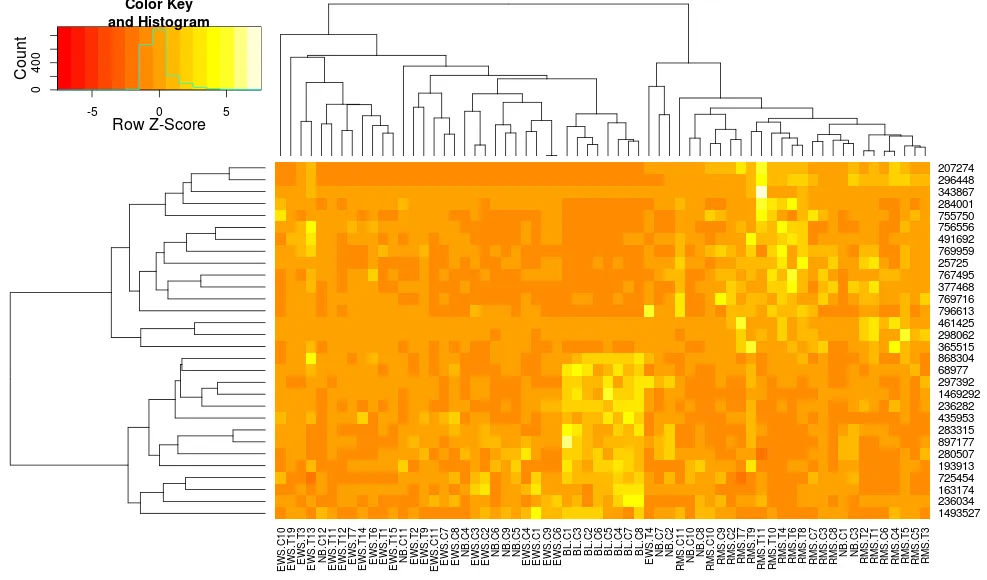
这张图片看起来并不完全一样,但更接近了。我如何使用heatmap.2重现heatplot的结果?它们之间有什么区别吗?
编辑2:关键的区别似乎在于heatplot使用以下方法对数据进行行列重新缩放:
if (dualScale) {
print(paste("Data (original) range: ", round(range(data),
2)[1], round(range(data), 2)[2]), sep = "")
data <- t(scale(t(data)))
print(paste("Data (scale) range: ", round(range(data),
2)[1], round(range(data), 2)[2]), sep = "")
data <- pmin(pmax(data, zlim[1]), zlim[2])
print(paste("Data scaled to range: ", round(range(data),
2)[1], round(range(data), 2)[2]), sep = "")
}
这是我尝试导入到我的
heatmap.2调用中的内容。我喜欢它的原因是它使低值和高值之间的对比更大,而仅仅将zlim传递给heatmap.2会被简单地忽略。如何在保留列聚类的同时使用这种“双重缩放”?我想要的只是增加对比度,就像这样:heatplot(..., dualScale=TRUE, scale="none"),与低对比度相比,你可以得到:heatplot(..., dualScale=FALSE, scale="row"),对此有什么想法吗?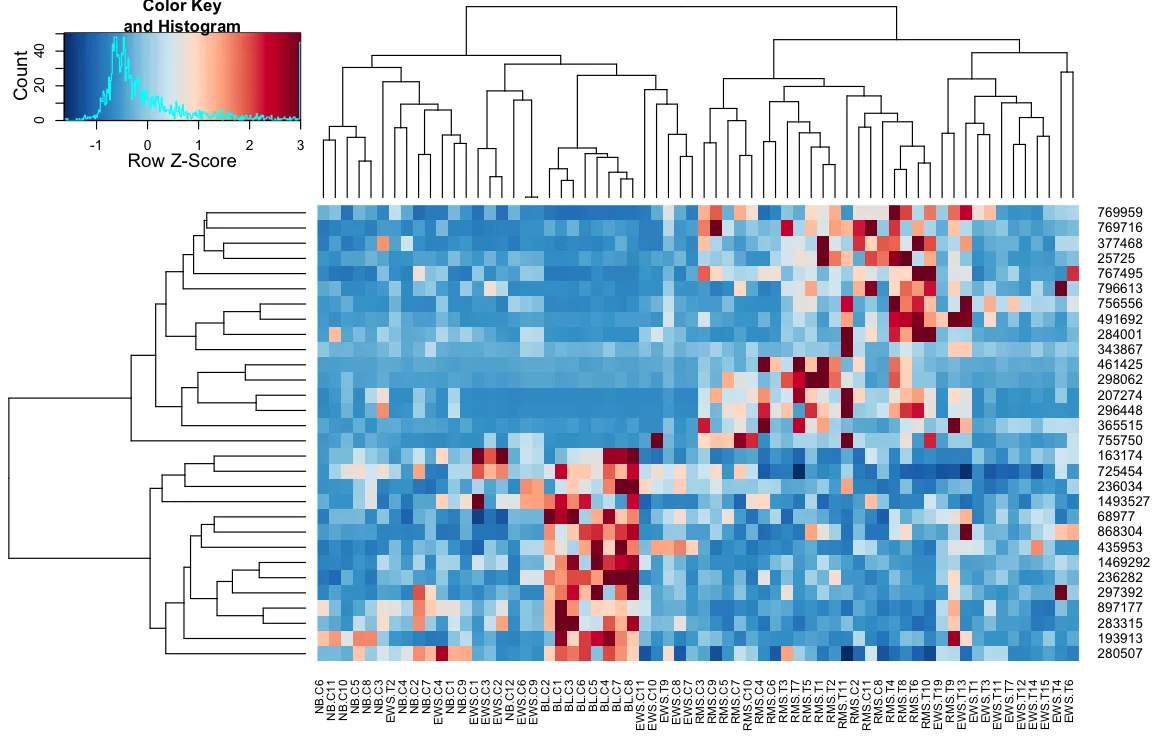
symbreaks=FALSEпјҢд»ҘиҺ·еҫ—зұ»дјјдәҺheatplotзҡ„зқҖиүІгҖӮдҪҶжҳҜеҲ—ж ‘зҠ¶еӣҫиҝҳйңҖиҰҒж”№иҝӣгҖӮ - harkmugsymbreaks的作用。关于列树状图上有何不同,您有什么想法吗? - user248237symbreaks=FALSE使得颜色不对称,就像在heatplot中看到的那样,0 值的颜色不是白色(仍然有点蓝色)。至于树状图,我认为heatmap.2可能做得更好。请注意,在heatmap.2中,EWS.T1 和 EWS.T6 是并排的,而在heatplot中,则是 EWS.T4 和 EWS.T6。前者的距离为 0.2,而后者的距离为 0.5。 - harkmugheatplot的一个bug吗? - user248237heatplot内使用的distEisen函数来解释吗?不幸的是,我想不到一种方法来传递相关距离给heatplot,因为它只接受作为dist()参数的内容,而dist()没有相关距离。如果heatplot()接受相关距离,那么这将是可能的。 - user248237