默认情况下,R的heatmap将对行和列进行聚类:
mtscaled = as.matrix(scale(mtcars))
heatmap(mtscaled, scale='none')
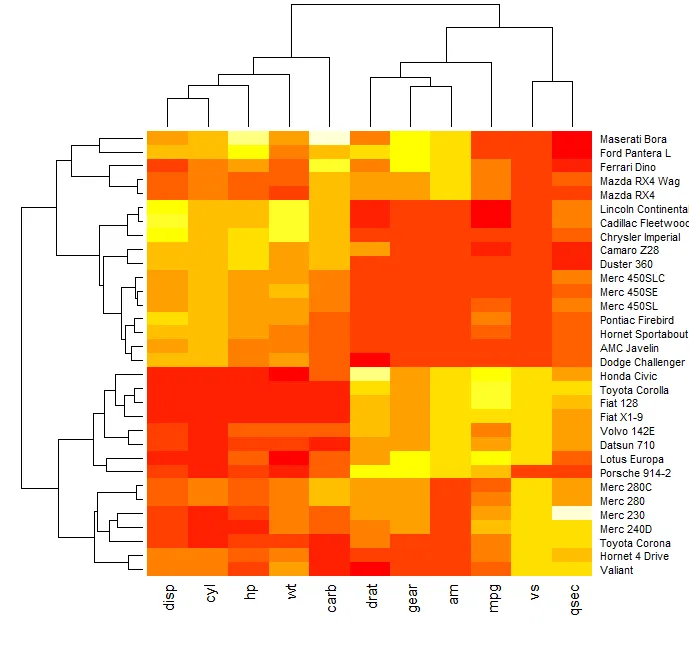
我可以禁用聚类:
heatmap(mtscaled, Colv=NA, Rowv=NA, scale='none')
然后树状图消失了: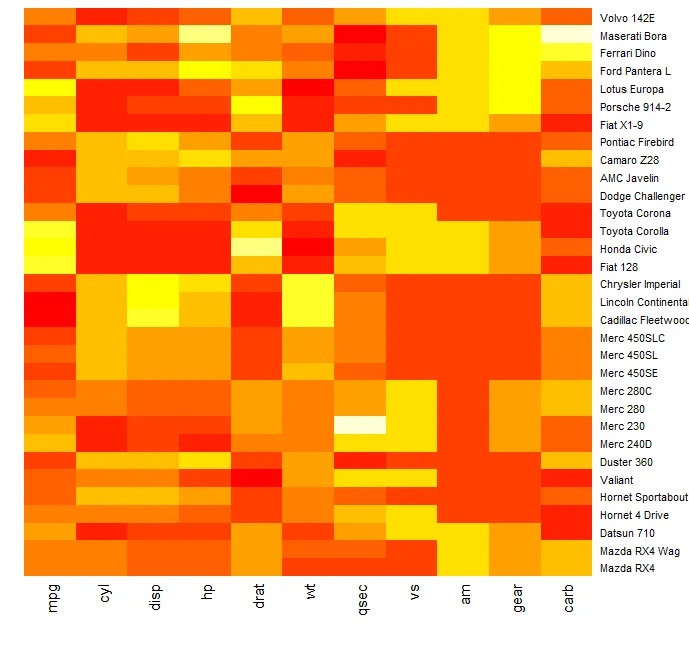
但是现在数据不再被聚类。
我不想显示树状图,但仍然希望对行和/或列进行聚类。我该怎么办?
我想要的示例: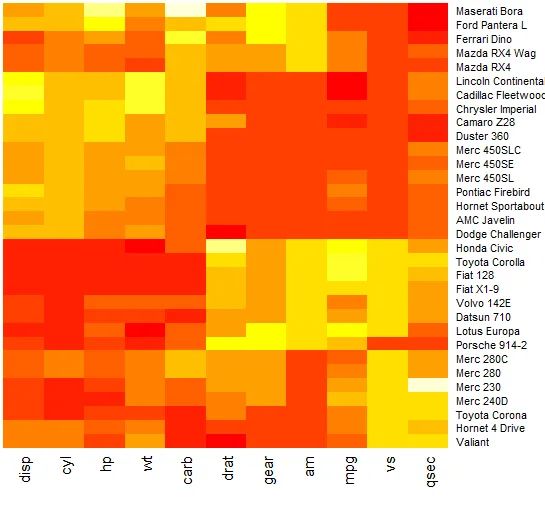
默认情况下,R的heatmap将对行和列进行聚类:
mtscaled = as.matrix(scale(mtcars))
heatmap(mtscaled, scale='none')

我可以禁用聚类:
heatmap(mtscaled, Colv=NA, Rowv=NA, scale='none')
然后树状图消失了:
但是现在数据不再被聚类。
我不想显示树状图,但仍然希望对行和/或列进行聚类。我该怎么办?
我想要的示例:
您可以使用pheatmap来完成此操作:
mtscaled <- as.matrix(scale(mtcars))
pheatmap::pheatmap(mtscaled, treeheight_row = 0, treeheight_col = 0)
查看 pheatmap 输出结果:
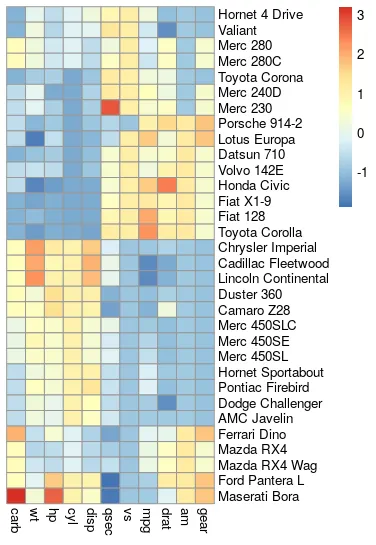
pheatmap默认进行聚类 (cluster_rows = TRUE, cluster_cols = TRUE)。 - Karel Fišerlibrary(gplots)
heatmap.2(mtscaled,dendrogram='none', Rowv=TRUE, Colv=TRUE,trace='none')
Rowv - 是TRUE,这意味着根据行均值计算和重新排列系统树。
Colv - 列应该与行被同等地处理。
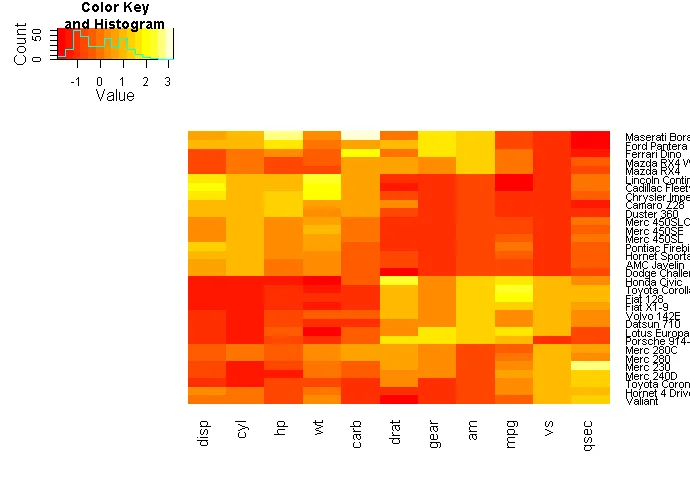
你可以依靠基本的R结构,考虑以下方法,通过自己构建hclust树来实现。
mtscaled = as.matrix(scale(mtcars))
row_order = hclust(dist(mtscaled))$order
column_order = hclust(dist(t(mtscaled)))$order
heatmap(mtscaled[row_order,column_order], Colv=NA, Rowv=NA, scale="none")
不需要安装额外的垃圾。
library(ComplexHeatmap)
Heatmap(as.matrix(iris[,1:4]), name = "mat", show_column_dend = FALSE, show_row_dend = FALSE)
library(pheatmap)
mtscaled = as.matrix(scale(mtcars))
H = pheatmap(mtscaled)
pheatmap(mtscaled[H$tree_row$order,H$tree_col$order],cluster_rows = F,cluster_cols = F)
使用基本的R热图函数两次绘制树状图。获取第一次运行的输出,该输出进行了聚类但必须绘制树状图,并将其再次输入到热图函数中。这一次,不进行聚类,也不绘制树状图。
#生成一个具有一定结构的随机对称矩阵,并制作热图
M100s<-matrix(runif(10000),nrow=100)
M100s[2,]<-runif(100,min=0.1,max=0.2)
M100s[4,]<-runif(100,min=0.1,max=0.2)
M100s[6,]<-runif(100,min=0.1,max=0.2)
M100s[99,]<-runif(100,min=0.1,max=0.2)
M100s[37,]<-runif(100,min=0.1,max=0.2)
M100s[lower.tri(M100s)] <- t(M100s)[lower.tri(M100s)]
heatmap(M100s)
#保存输出结果
OutputH <- heatmap(M100s)
#再次运行,不使用聚类或树状图
M100c <- M100s
M100c1 <- M100c[,OutputH$rowInd]
M100c2 <- M100c1[OutputH$colInd,]
heatmap(M100c2,Rowv = NA, Colv = NA, labRow = NA, labCol = NA)