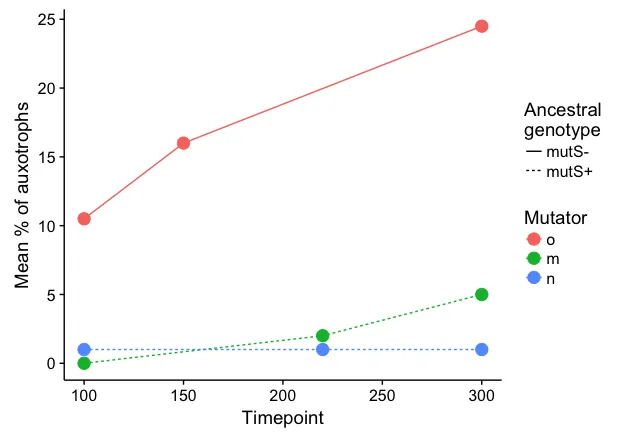
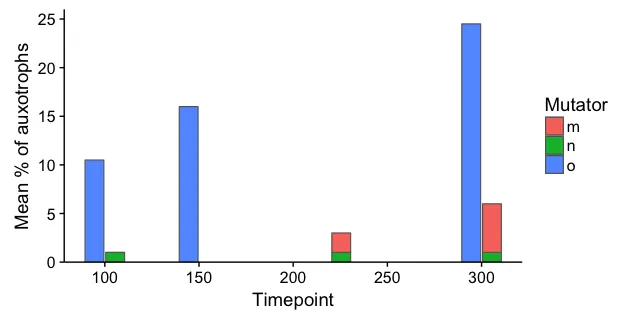
我正在尝试重新创建这个图表,但不想使用可怕的3D条形图和不清晰的X轴(这些是不同的时间点,很难确定它们的时间)。
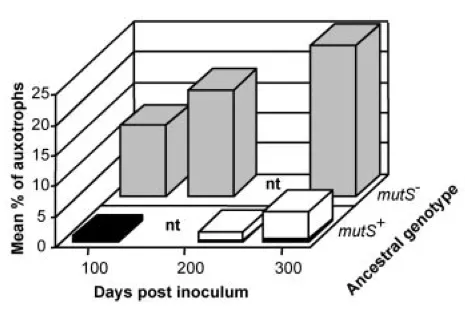
(来自Science 291,第5513期(2001年):2606-8,除此之外是一篇很好的论文。)
我的第一反应是做类似于他们所做的事情,使用2D条形图和不同的x轴标签,对基因型使用dodged bars,然后使用堆积条形图在前面的条形图上获得黑白分割,但是这里有几个其他好问题说你不能这样做。
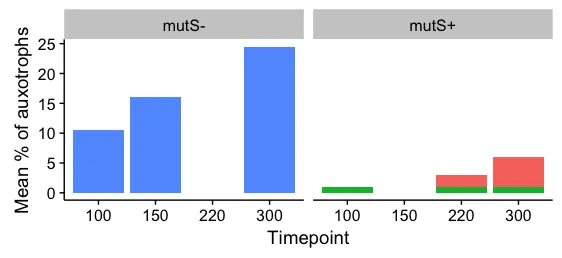
我的下一个方法是使用faceting(以下是代码),这个方法效果还不错,但我很想看到更好的方法来做这个。有没有一种方法可以将某些变量堆叠起来,而将其他变量闪避掉?或者只是一般做法更好吗?
编辑:为了澄清,我认为显示堆叠条形图的总数很重要(在这种情况下是m和n,最初是黑色和白色),因为这代表了一个测量数量,然后分割是另一个测量。
library(tidyverse)
library(cowplot)
data = tribble(
~Timepoint, ~`Ancestral genotype`, ~Mutator, ~`Mean % of auxotrophs`,
100, 'mutS-', 'o', 10.5,
150, 'mutS-', 'o', 16,
220, 'mutS-', 'o', NA,
300, 'mutS-', 'o', 24.5,
100, 'mutS+', 'n', 1,
150, 'mutS+', 'n', NA,
220, 'mutS+', 'n', 1,
300, 'mutS+', 'n', 1,
100, 'mutS+', 'm', 0,
150, 'mutS+', 'm', NA,
220, 'mutS+', 'm', 2,
300, 'mutS+', 'm', 5
)
data <- data %>% mutate(Timepoint = as.character(Timepoint))
data %>% ggplot(aes(x = Timepoint, y = `Mean % of auxotrophs`)) +
geom_col(aes(fill = Mutator), position = 'stack') + facet_grid(~`Ancestral genotype` ) +
guides(fill=FALSE)