我正在研究一些不同场景下的线性回归示例,比较使用 Normalizer 和 StandardScaler 的结果,并且结果令人困惑。
我正在使用波士顿住房数据集,并准备好这样做:
import numpy as np
import pandas as pd
from sklearn.datasets import load_boston
from sklearn.preprocessing import Normalizer
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LinearRegression
#load the data
df = pd.DataFrame(boston.data)
df.columns = boston.feature_names
df['PRICE'] = boston.target
我目前正在试图理解以下场景得到的结果:
- 使用参数
normalize=True初始化线性回归 vs 使用Normalizer - 使用参数
fit_intercept=False初始化线性回归并进行标准化与不标准化比较。
总的来说,我觉得结果令人困惑。
这是我设置一切的方法:
# Prep the data
X = df.iloc[:, :-1]
y = df.iloc[:, -1:]
normal_X = Normalizer().fit_transform(X)
scaled_X = StandardScaler().fit_transform(X)
#now prepare some of the models
reg1 = LinearRegression().fit(X, y)
reg2 = LinearRegression(normalize=True).fit(X, y)
reg3 = LinearRegression().fit(normal_X, y)
reg4 = LinearRegression().fit(scaled_X, y)
reg5 = LinearRegression(fit_intercept=False).fit(scaled_X, y)
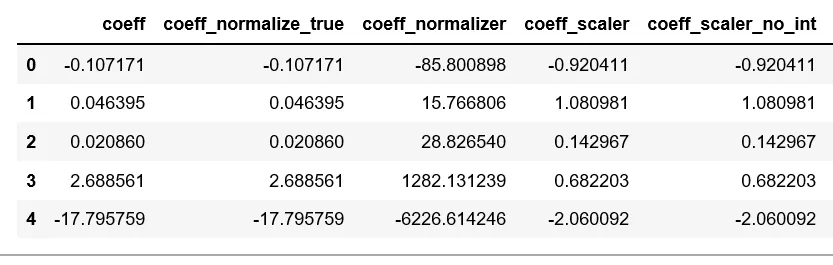
然后,我创建了3个单独的数据框来比较每个模型的R分数、系数值和预测结果。
为了创建用于比较每个模型系数值的数据框,我执行了以下操作:
#Create a dataframe of the coefficients
coef = pd.DataFrame({
'coeff': reg1.coef_[0],
'coeff_normalize_true': reg2.coef_[0],
'coeff_normalizer': reg3.coef_[0],
'coeff_scaler': reg4.coef_[0],
'coeff_scaler_no_int': reg5.coef_[0]
})
这是我创建数据帧以比较每个模型的 R^2 值的方法:
scores = pd.DataFrame({
'score': reg1.score(X, y),
'score_normalize_true': reg2.score(X, y),
'score_normalizer': reg3.score(normal_X, y),
'score_scaler': reg4.score(scaled_X, y),
'score_scaler_no_int': reg5.score(scaled_X, y)
}, index=range(1)
)
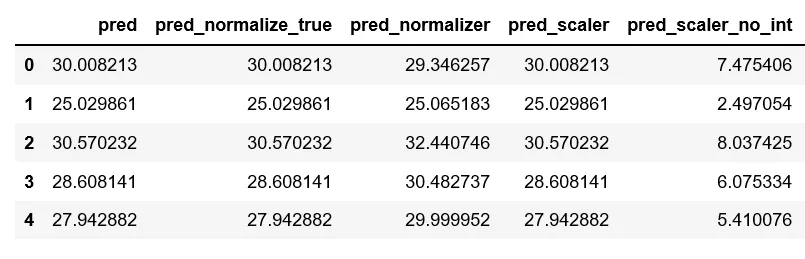
最后,这是一个比较每个预测结果的数据框:
predictions = pd.DataFrame({
'pred': reg1.predict(X).ravel(),
'pred_normalize_true': reg2.predict(X).ravel(),
'pred_normalizer': reg3.predict(normal_X).ravel(),
'pred_scaler': reg4.predict(scaled_X).ravel(),
'pred_scaler_no_int': reg5.predict(scaled_X).ravel()
}, index=range(len(y)))
这是生成的数据框:
我有三个问题无法解决:
- 为什么前两个模型完全没有区别?看起来设置
normalize=False没有任何作用。我可以理解预测值和R ^ 2值相同,但我的特征具有不同的数字比例,因此我不确定归一化为什么根本没有影响。当您考虑使用StandardScaler时,这种情况变得更加令人困惑,因为它会显着改变系数。 - 我不明白为什么使用
Normalizer模型与其他模型相比,系数值会有如此激烈的不同,特别是当使用LinearRegression(normalize=True)模型时根本没有变化。
如果您查看每个文档,则似乎它们非常相似,甚至相同。
在sklearn.linear_model.LinearRegression()的文档中,写道:
normalize:布尔型,可选,默认为False
当fit_intercept设置为False时,将忽略此参数。如果为True,则回归器X将在回归之前通过减去平均值并除以l2范数进行规范化。
同时,sklearn.preprocessing.Normalizer的文档指出默认情况下规范化为l2范数。
我不见得这两个选项有什么区别,而且我也不明白为什么一个会与另一个具有如此激烈的系数差异。
- 使用
StandardScaler的模型结果对我来说是连贯的,但我不理解使用StandardScaler和设置set_intercept=False的模型为什么表现如此糟糕。
从线性回归模块的文档中得知:
fit_intercept : 布尔值,可选参数,默认为True
是否计算此模型的截距。如果设置为False,则在计算中不使用截距(例如,数据已预期为已经居中)。
StandardScaler用于居中数据,所以我不明白为什么将其与fit_intercept=False一起使用会产生不连贯的结果。

std = StandardScaler() std.fit(X.values) X_tr = std.transform(X.values)的目的是什么?之后,我们可以安全地运行套索模型并使用回归系数来选择预测变量吗? - Edison