我实现了一个函数,用于将`np.dot`应用于从内存映射数组中明确读入核心内存的块:
import numpy as np
def _block_slices(dim_size, block_size):
"""Generator that yields slice objects for indexing into
sequential blocks of an array along a particular axis
"""
count = 0
while True:
yield slice(count, count + block_size, 1)
count += block_size
if count > dim_size:
raise StopIteration
def blockwise_dot(A, B, max_elements=int(2**27), out=None):
"""
Computes the dot product of two matrices in a block-wise fashion.
Only blocks of `A` with a maximum size of `max_elements` will be
processed simultaneously.
"""
m, n = A.shape
n1, o = B.shape
if n1 != n:
raise ValueError('matrices are not aligned')
if A.flags.f_contiguous:
max_cols = max(1, max_elements / m)
max_rows = max_elements / max_cols
else:
max_rows = max(1, max_elements / n)
max_cols = max_elements / max_rows
if out is None:
out = np.empty((m, o), dtype=np.result_type(A, B))
elif out.shape != (m, o):
raise ValueError('output array has incorrect dimensions')
for mm in _block_slices(m, max_rows):
out[mm, :] = 0
for nn in _block_slices(n, max_cols):
A_block = A[mm, nn].copy()
out[mm, :] += np.dot(A_block, B[nn, :])
del A_block
return out
我随后进行了一些基准测试,将我的
blockwise_dot函数与直接应用于内存映射数组的普通
np.dot函数进行比较(请参见以下基准测试脚本)。 我使用的是针对OpenBLAS v0.2.9.rc1(从源代码编译)的numpy 1.9.0.dev-205598b。该机器是运行Ubuntu 13.10的四核笔记本电脑,具有8GB RAM和SSD,并且已禁用交换文件。
结果
正如@Bi Rico所预测的那样,计算点积所需的时间与
A的维数美妙地成为
O(n)。在缓存的
A块上操作比仅调用整个内存映射数组上的普通
np.dot函数会大大提高性能:
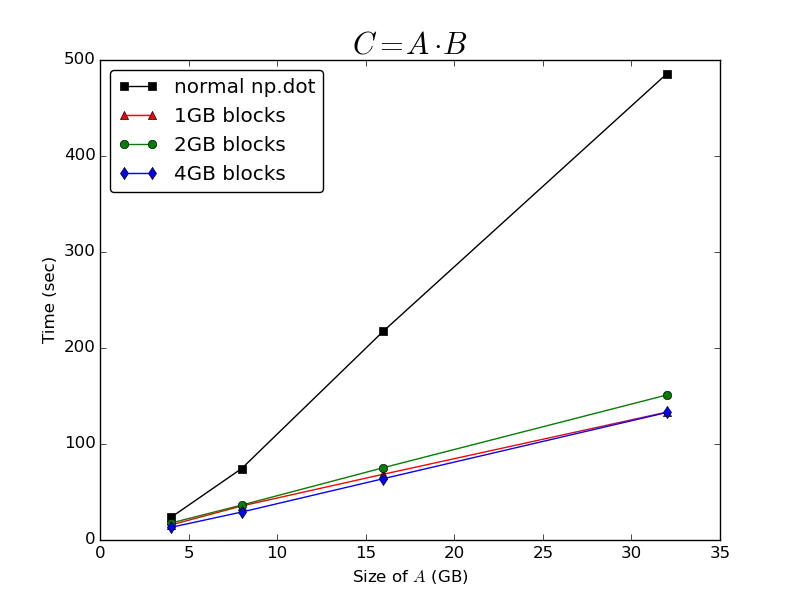
它对正在处理的块的大小非常不敏感-在以1GB、2GB或4GB块处理数组所需的时间之间几乎没有什么区别。 我得出结论,无论
np.memmap数组本地实现了什么缓存,它似乎都非常次优于计算点积。
进一步的问题
手动实现此缓存策略仍然有点麻烦,因为我的代码可能必须在具有不同物理内存量和潜在不同操作系统的机器上运行。出于这个原因,我仍然对是否有方法控制内存映射数组的缓存行为以提高
np.dot的性能感兴趣。
当我运行基准测试时,我注意到了一些奇怪的内存处理行为-当我在整个
A上调用
np.dot时,我从未看到我的Python进程的常驻集大小超过约3.8GB,即使我有大约7.5GB的可用RAM。这导致我怀疑对于一个
np.memmap数组,可能会对其允许占用的物理内存量施加一些限制-我之前认为它将使用操作系统允许它获取的任何RAM。在我的情况下,能够增加此限制可能非常有益。
是否有人对
np.memmap数组的缓存行为有更深入的了解,可以帮助解释这一点?
基准测试脚本
def generate_random_mmarray(shape, fp, max_elements):
A = np.memmap(fp, dtype=np.float32, mode='w+', shape=shape)
max_rows = max(1, max_elements / shape[1])
max_cols = max_elements / max_rows
for rr in _block_slices(shape[0], max_rows):
for cc in _block_slices(shape[1], max_cols):
A[rr, cc] = np.random.randn(*A[rr, cc].shape)
return A
def run_bench(n_gigabytes=np.array([16]), max_block_gigabytes=6, reps=3,
fpath='temp_array'):
"""
time C = A * B, where A is a big (n, n) memory-mapped array, and B and C are
(n, o) arrays resident in core memory
"""
standard_times = []
blockwise_times = []
differences = []
nbytes = n_gigabytes * 2 ** 30
o = 64
max_elements = int((max_block_gigabytes * 2 ** 30) / 4)
for nb in nbytes:
n = int(np.sqrt(nb / 4))
with open(fpath, 'w+') as f:
A = generate_random_mmarray((n, n), f, (max_elements / 2))
B = np.random.randn(n, o).astype(np.float32)
print "\n" + "-"*60
print "A: %s\t(%i bytes)" %(A.shape, A.nbytes)
print "B: %s\t\t(%i bytes)" %(B.shape, B.nbytes)
best = np.inf
for _ in xrange(reps):
tic = time.time()
res1 = np.dot(A, B)
t = time.time() - tic
best = min(best, t)
print "Normal dot:\t%imin %.2fsec" %divmod(best, 60)
standard_times.append(best)
best = np.inf
for _ in xrange(reps):
tic = time.time()
res2 = blockwise_dot(A, B, max_elements=max_elements)
t = time.time() - tic
best = min(best, t)
print "Block-wise dot:\t%imin %.2fsec" %divmod(best, 60)
blockwise_times.append(best)
diff = np.linalg.norm(res1 - res2)
print "L2 norm of difference:\t%g" %diff
differences.append(diff)
del A, B
del res1, res2
os.remove(fpath)
return (np.array(standard_times), np.array(blockwise_times),
np.array(differences))
if __name__ == '__main__':
n = np.logspace(2,5,4,base=2)
standard_times, blockwise_times, differences = run_bench(
n_gigabytes=n,
max_block_gigabytes=4)
np.savez('bench_results', standard_times=standard_times,
blockwise_times=blockwise_times, differences=differences)

np.einsum,因为我想不出它比np.dot更快的特定原因。对于计算两个在核心内存中的数组的点积,np.dot将比等效的np.einsum调用更快,因为它可以使用更高度优化的BLAS函数。在我的情况下,几乎没有区别,因为我受到I/O限制。2)不,正如我在描述中所说的,它们是密集的矩阵。 - ali_m