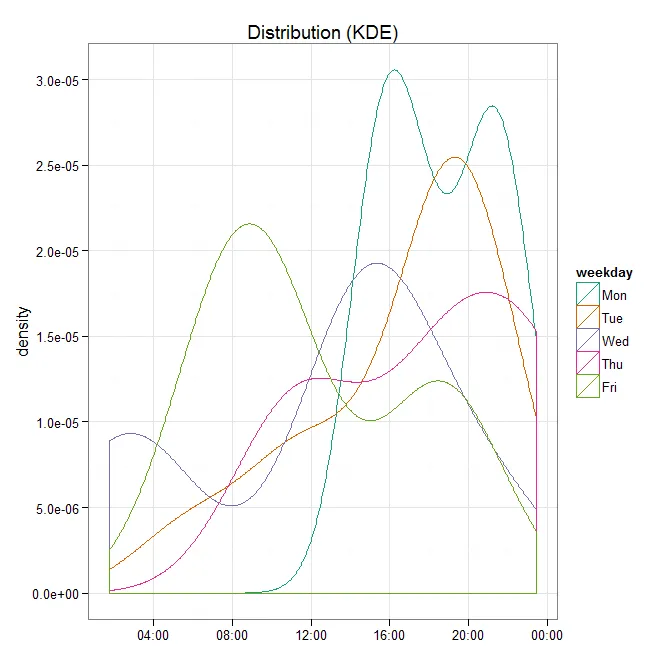
我正在尝试了解我的数据在一周中每小时的频率概述。因此,我将不同的日期压缩成一个单一的日子,这样只有时间不同,并添加了一个列来表示星期几作为一个有序因素。
以下是我的数据摘录:
my.log <- structure(list(Prorated = structure(c(1339535400, 1339536540, 1339524540, 1339480320, 1339537920, 1339529580, 1339500780, 1339532820, 1339522020, 1339522680, 1339465560, 1339529940, 1339472880, 1339508520, 1339519620, 1339536000, 1339526580, 1339514940, 1339518060, 1339512420, 1339513080, 1339500120, 1339543620, 1339485660, 1339496280, 1339526520, 1339514820, 1339531800, 1339531860, 1339501320), class = c("POSIXct", "POSIXt"), tzone = "%Y-%m-%d %H:%M:%S"), Wday = structure(c(1, 1, 1, 2, 1, 2, 2, 2, 2, 2, 3, 2, 3, 3, 3, 3, 4, 1, 1, 3, 3, 4, 4, 5, 5, 5, 1, 2, 2, 2), .Label = c("Mon", "Tue", "Wed", "Thu", "Fri", "Sat", "Sun"), class = c("ordered", "factor"))), .Names = c("Prorated", "Wday"), row.names = c(NA, 30), class = "data.frame")
range(my.log$Prorated)
# here (n = 30):
# [1] "2012-06-12 01:46:00" "2012-06-12 23:27:00"
# w/ full data set (n = approx. 75000):
# [1] "2012-06-12 00:00:00" "2012-06-12 23:59:00"
当我现在尝试使用以下代码绘制密度图时...
library("ggplot2")
library("scales")
p <- ggplot(my.log) + theme_bw() +
geom_density(aes(Prorated, colour=Wday)) +
scale_color_brewer("weekday", palette="Dark2") +
scale_x_datetime("", breaks=date_breaks("4 hours"),
labels=date_format("%H:00")) +
opts(title="Distribution (KDE)")
print(p)
因为两个数据集的X轴并不从00:00开始,而是从02:00开始,导致整个密度图被移到了下一天。(我想在这里发布一张图片,但由于我是SO的新手,所以无法这样做。您可以在ImageShack找到它。)
因此,我的问题是:是否有选项告诉qqplot()应该从00:00开始绘制密度图?
我查看了SO的相关问题(或回答),但没有找到任何答案。我所能想到的唯一选择是xlim()或scale_x_continuous(limits=...)。但据我所知,这两种方法都不适用于这里。
前者会删除数据点(或者不会,因为输入数据框中的所有数据已经在正确的范围内),而后者只会移动视角,并因此在23:59处切断图形,而不添加这些(现在隐藏的)数据点在开头...所以当我使用
scale_x_datetime("", breaks=date_breaks("4 hours"), labels=date_format("%H:00"),
limits=c(as.POSIXct("2012-06-12 00:00:00"), as.POSIXct("2012-06-12 23:59:00"))
在上面的代码中,图形看起来不正确/未显示所有数据。