我使用ggplot2绘制鸢尾花数据图。看起来ggplot2会自动将数据标准化到(0.1)区间。如何在不进行任何标准化处理的情况下绘制数据图?
library(ggplot2)
p <- ggpcp(iris, vars = names(iris[1:4]))
p + geom_line(aes(color = Species)) + ylim(0,8)
我不是以英语为母语的人,并且很抱歉会造成歧义。实际上,虹膜数据从0到8变化不等。我想要绘制反映真实值而非标准化值(将原始数据转换为(0,1)区间)的数据图。
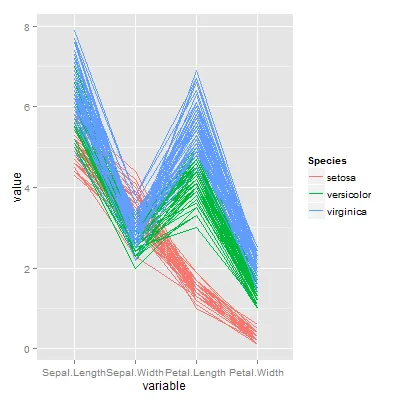
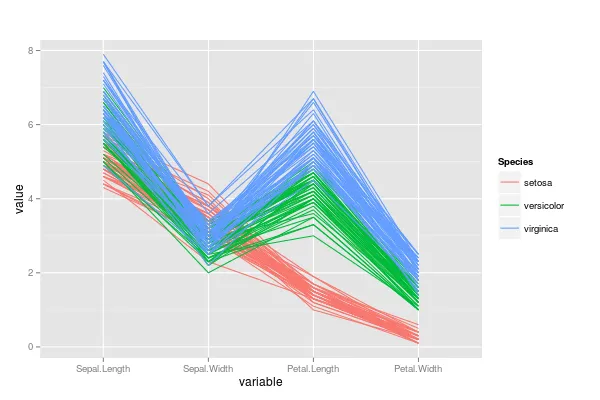
ggpcp()函数有一个scale = FALSE的选项,但后来因为它依赖于“reshape”包而被删除了。 - A5C1D2H2I1M1N2O1R2T1geom_path中设置alpha=0.2,则图表不会那么拥挤。 - csgillespie