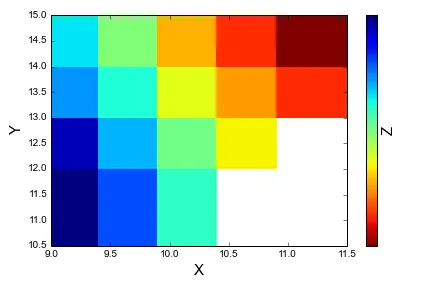
如何告诉interp2d忽略NaN值?
我有一个带有任意值z的表面x和y。
x = np.array([[9.19632, 9.62141, 10.0829, np.isnan, np.isnan],
[9.21164, 9.64347, 10.1392, 10.5698, np.isnan],
[9.22175, 9.65439, 10.1423, 10.6301, 11.0323],
[9.21632, 9.67060, 10.1474, 10.6230, 11.0818]])
y = np.array([[11.5466,11.6485,11.7619, np.isnan, np.isnan],
[12.4771, 12.5460, 12.5453, 12.7142, np.isnan],
[13.5578, 13.5581, 13.5505, 13.5309, 13.6081],
[14.5653, 14.5504, 14.5036, 14.5145, 14.5060]])
z = np.array([[0.466113, 0.0484404, -0.385355, np.isnan, np.isnan],
[0.366125, -0.160165, -0.548668, -0.888301,np.isnan],
[-0.0970777, -0.346734, -0.826576, -1.08412, -1.33129],
[-0.259981, -0.586938, -1.03477, -1.32384, -1.61500]])
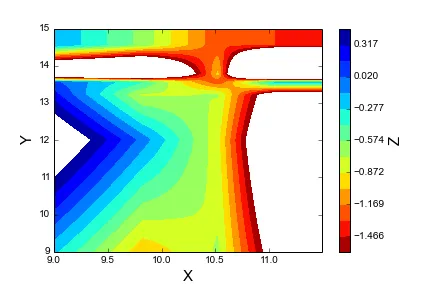
我已经使用掩膜数组生成了上述颜色网格,但是当我尝试使用二维插值创建更精细的网格时失败了。下面是我的代码,注意我将nan值设置为零以获得这个结果,因此它显然会破坏“正确”的插值。相反,我想忽略它们并留空参数空间。
f = interp.interp2d(x,y,z, kind='linear')
xnew = np.arange(9,11.5, 0.01)
ynew = np.arange(9,15, 0.01)
znew = f(xnew, ynew)
levels = np.linspace(zmin, zmax, 15)
plt.ylabel('Y', size=15)
plt.xlabel('X', size=15)
cmap = plt.cm.jet_r
cmap.set_bad('white',0.1) # set nan to white
cs = plt.contourf(xnew, ynew, znew, levels=levels, cmap=cmap)
cbar = plt.colorbar(cs)
cbar.set_label('Z', rotation=90, fontsize=15) # gas fraction
plt.show()
我希望能够简单地创建一个平滑的颜色图,其中由x和y界定的区域根据z进行着色。
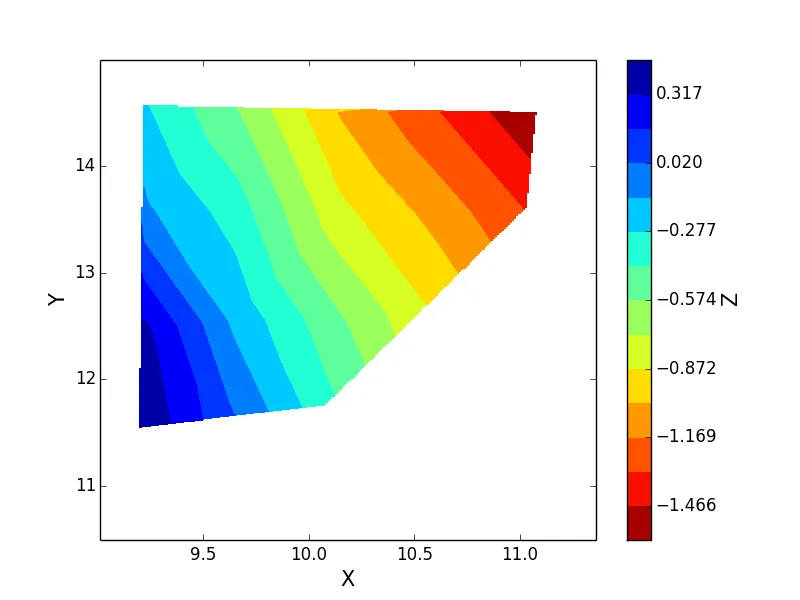
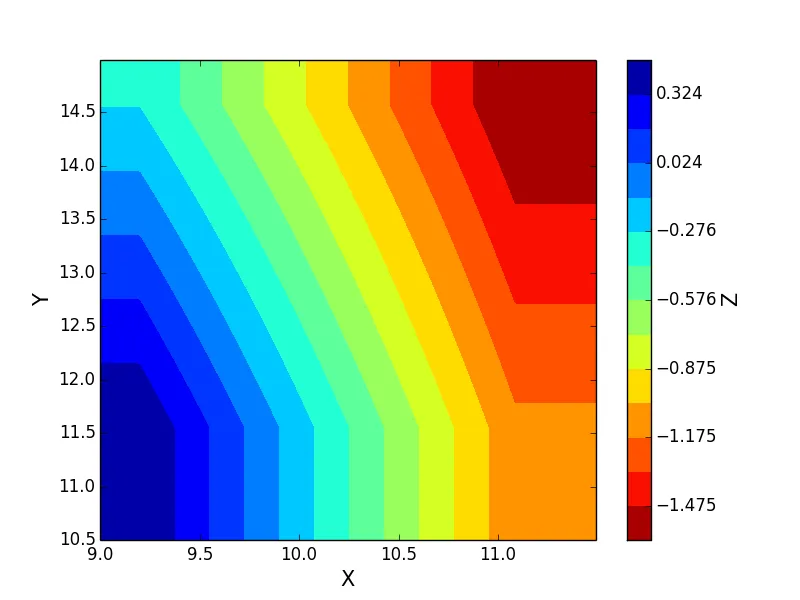
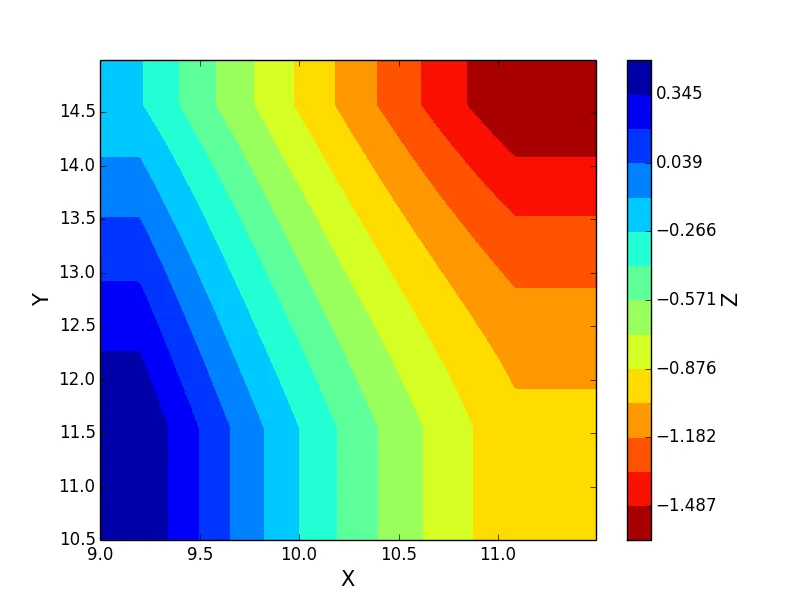
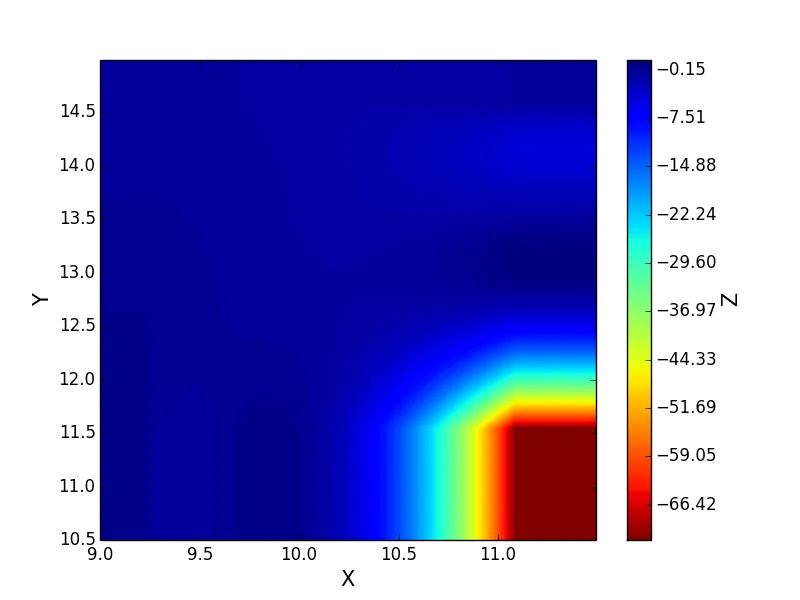
SmoothBivariateSpline进行外推的图像。 - Noel Segura MerazSmoothBivariateSpline类呢?感谢你的帮助。 - smashbronan?我认为griddata可以处理它们。 - FooBargriddata可以处理函数插值中的nan值,但不能作为坐标点。由于原始问题中也出现了nan作为坐标,因此将它们全部删除更容易一些。 - Noel Segura Meraz