我有一组简单的方向关系(父->子)想要绘制。我的数据结构是这样的,有许多离散的子网络。以下是一个类似于我的假数据。
require(igraph)
parents<-c("A","A","C","C","F","F","H","I")
children<-c("B","C","D","E","G","H","I","J")
begats<-data.frame(parents=parents,children=children)
graph_begats<-graph.data.frame(begats)
plot(graph_begats)
假数据中有两个不同的子网络,每个子网络都是严格的父-子血统。我需要在同一个窗口中(理想情况下使用相同的顶点坐标系统)将这两个血统作为树形网络绘制出来。我尝试使用layout.reingold.tilford(),但最多只能绘制一棵树,所有其他顶点都绘制在根顶点之上,如下所示。
lo<-layout.reingold.tilford(graph_begats,root=1)
plot(graph_begats,layout=lo)
有没有关于对任意数量的离散谱系进行此操作的想法?
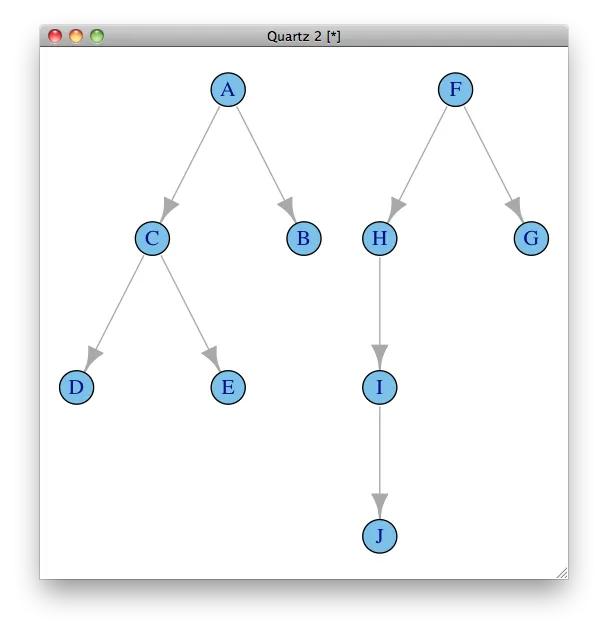
clusters()或decompose.graph()将网络拆分,然后为每个部分单独计算布局,最后通过移动一个布局矩阵将它们合并。 - Gabor Csardidecompose.graph()就是我需要的。矩阵移位还在努力中,但我正在接近目标。 - Andrew Barrlayout.reingold.tilford()获取树形布局,我需要一个识别根节点的方法。我通过采用topological sort()函数返回的第一个顶点来实现这一点,如topological.sort(theGraph)[1]所示。这在我的示例数据中的子图中并不是必需的,但通常在真实数据中是必需的。 - Andrew Barr