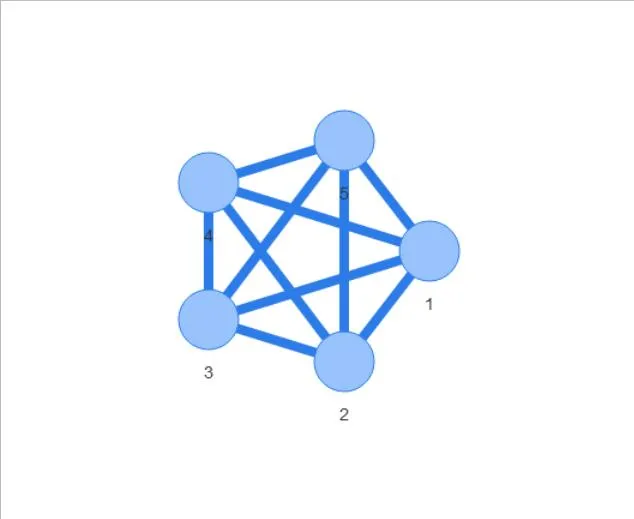
关于将igraph与visNetwork结合使用,我有一个非常简单的问题。我想用visEdges(value=E(graph)$weight)给边加权,但这并不起作用。 这里是一个玩具示例,以说明这个问题:
test
[,1] [,2] [,3] [,4] [,5]
[1,] 0 1 3 7 1
[2,] 4 0 8 9 5
[3,] 10 3 0 8 3
[4,] 5 1 5 0 7
[5,] 8 2 7 4 0
library(igraph); library(visNetwork)
test.gr <- graph_from_adjacency_matrix(test, mode="undirected", weighted=T)
如果我现在尝试将其可视化为加权图,它不会绘制出来:
test.gr %>%
visIgraph(layout = "layout_in_circle") %>%
visEdges(value = E(test.gr)$weight)
如果我使用
test.gr %>%
visIgraph(layout = "layout_in_circle") %>%
visEdges(value = 10)
相反,我得到了一个图:
但这当然不是我想要的。我希望根据E(test.gr)$weight获得不同的边缘宽度。
您能告诉我如何做到这一点吗?