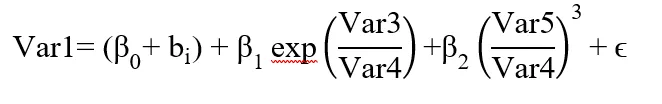我正在使用混合效应模型,由于我的方法论的特殊性,我需要解决下面模型的积分,然后绘制估计值的图形。
换句话说,我需要解决下面的积分: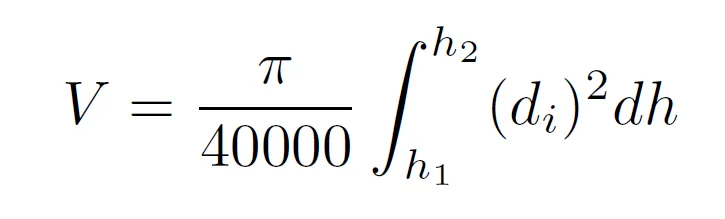 其中,
其中,
在我所处的问题的文献中,很少有人使用混合效应模型来达到这个目的,绝大多数只使用简单线性回归模型。然而,对于我的问题,必须使用混合模型。
该模型的定义如下:
换句话说,我需要解决下面的积分:
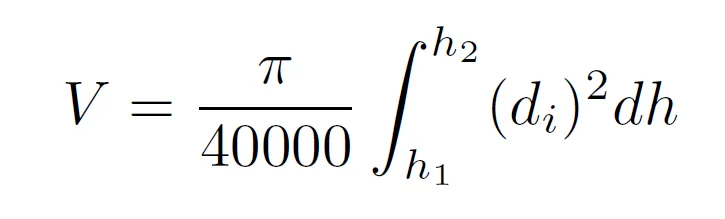 其中,
其中,di^2 是我的模型中的 Var3,而 dh 是对应于混合效应模型的函数。在我所处的问题的文献中,很少有人使用混合效应模型来达到这个目的,绝大多数只使用简单线性回归模型。然而,对于我的问题,必须使用混合模型。
该模型的定义如下:
在考虑变量Var2的情况下,随机效应bi被引入截距中。
只考虑模型的固定部分,即固定效应模型,我执行的解决积分的过程如下:
数据:https://drive.google.com/file/d/1hFb1OPO0jxQw7_u62swnkRXbOH81ygDD/view?usp=sharing
很抱歉要将数据放在链接中,但是我找不到一个内部的R数据库,可以匹配我的问题。
fitmixedmodel <- lme(log(Var1)~I(exp(Var3/Var4))+
(I((Var5/Var4)^3)),
random = ~1|Var2,
dados, method="REML")
summary(fitmixedmodel)
volume <- dados[dados$Var5 == 0.1,]
fmixedmodel <- function(Var3, Var5, Var4){
(pi/40000)*(Var3^2)*(coefficients(summary(fitmixedmodel))[1] +
coefficients(summary(fitmixedmodel))[2]*I(exp(Var3/Var4)) +
coefficients(summary(fitmixedmodel))[3]*(I((Var5/Var4)^3)))
}
vmixedmodel <- function(Var3, Var5, Var4){
integrate(Vectorize(fmixedmodel), lower = 0.1, upper = Var4, Var3 = Var3, Var4 = Var4)$value
}
mixed.vol <- mapply(FUN = vmixedmodel,
Var5 = as.list(volume$Var5),
Var3 = as.list(volume$Var3),
Var4 = as.list(volume$Var4))
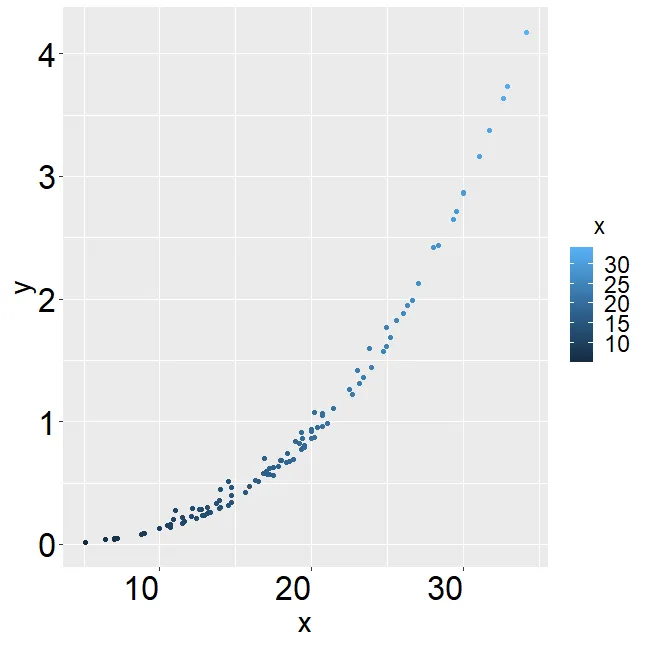
因此我得到了以下图表。
然而,请注意,计算此积分时我从未声明随机效应,即我仅从固定部分积分函数,也没有考虑随机部分。我该如何解决这个问题,即实际集成调整的混合模型方程?